Deregulierung diverser biochemischer Signalwege durch modRNA Injektionen Teil 2
Synthetic mRNA Vaccines and Transcriptomic Dysregulation: Evidence from New-Onset Adverse Events and Cancers Post-Vaccination
Streng genommen nicht neu. Ein Paper, welches das zeigt, erschien schon im Januar, nur interpretieren die Autoren das Januar Papers ihre Daten nicht entsprechend, auch wenn die Datenlage klar ist.
Deregulierung diverser biochemischer Signalwege durch modRNA Injektionen
Im Januar erschien ein Paper, welches sich endlich ein wenig mit den biochemischen Folgen durch die modRNA Plörren beschäftigt.
Nun eine zweite Publikation, die das obige Paper bestätigt, aber von Autoren ist, die vor Gericht Nasenrümpfen hervorrufen dürften, weil sie eben offensichtlich zum Widerstand gehören. Zudem ist das Paper aktuell nur ein Preprint.
von Ranke, N., Zhang, W., Anokin, P., Shao, D., Bereimipour, A., Vu, M., Hulscher, N., McKernan, K. J., McCullough, P. A., & Catanzaro, J. A. (2025). Synthetic mRNA Vaccines and Transcriptomic Dysregulation: Evidence from New-Onset Adverse Events and Cancers Post-Vaccination. Preprints. https://doi.org/10.20944/preprints202507.2155.v1, https://www.preprints.org/manuscript/202507.2155/v1
Hickey et al.1 haben mit Microarrays gearbeitet, hier hat man mit Transkriptom Analyse auf RNA-Sequenz Basis gearbeitet. Zwei unterschiedliche Methoden, ähnliches desaströses Ergebnis.
“Transkriptomweite Differentialexpressionsprofile bei Personen, die von Impfstoffen betroffen sind. (A) Vulkan-Diagramm, das die differentielle Genexpression bei Personen mit neu auftretenden unerwünschten Ereignissen nach einer mRNA-COVID-19-Impfung (n = 3) im Vergleich zu gesunden Kontrollpersonen (n = 803) zeigt. (B) Vulkan-Diagramm, das die unterschiedliche Genexpression bei Personen zeigt, bei denen kurz nach der mRNA-COVID-19-Impfung neu auftretender Krebs diagnostiziert wurde (n = 7), im Vergleich zur gleichen Kontrollkohorte. Jeder Punkt steht für ein einzelnes Gen, das anhand der log₂-Fold-Change (x-Achse) und des –log₁₀-bereinigten p-Werts (y-Achse) dargestellt ist. Gene mit signifikanter Hochregulation (log₂FC > 1, padj < 0,05) sind rot markiert, während Gene mit signifikanter Herunterregulation (log₂FC < –1, padj < 0,05) blau dargestellt sind. Nicht signifikante Gene erscheinen in Grau. Diese Diagramme zeigen eine weit verbreitete transkriptionelle Dysregulation in beiden Patientengruppen und dienen als Grundlage für die anschließende Pathway-Anreicherungsanalyse.”
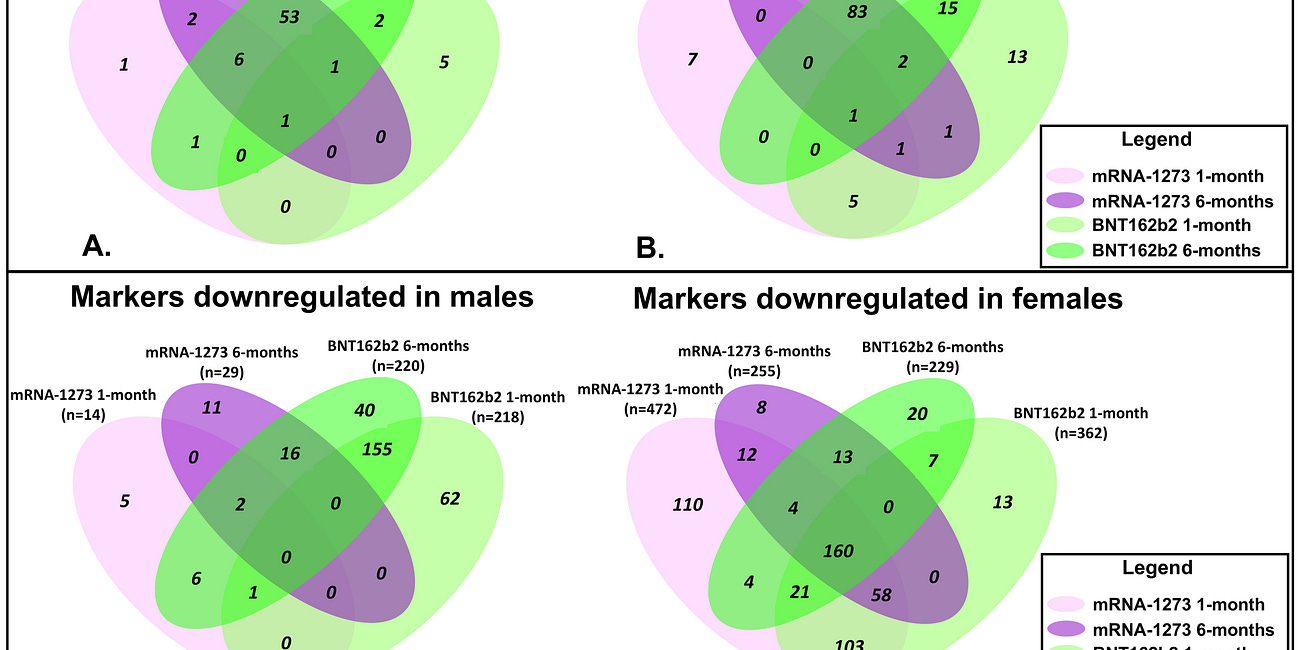
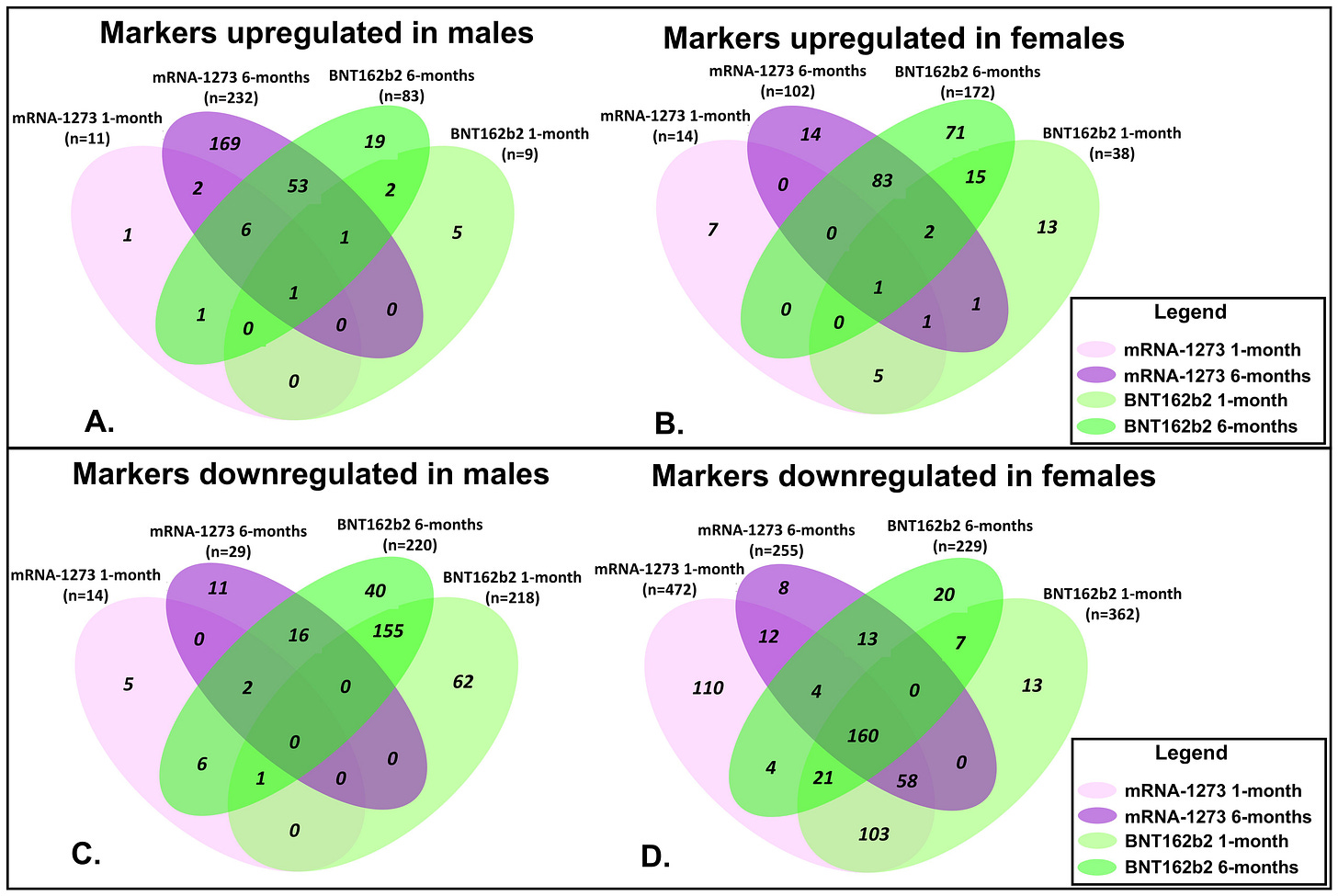
Hickey et al stellen das Ganze als Venn-Diagramme dar, haben aber anders als van Rake et al. keine Ungeimpften als Kontrolle. Das ist im neuen Preprint besser. Dafür haben Hickey et al deutlich mehr Probanden in der geimpften Gruppe. Da zeigt sich die Zugehörigkeit zur Informationsblase im Zugriff auf die Patientenpopulation. Der Widerstand hat einen ungeimpften Bias, der Systemzweig einen geimpften Bias.
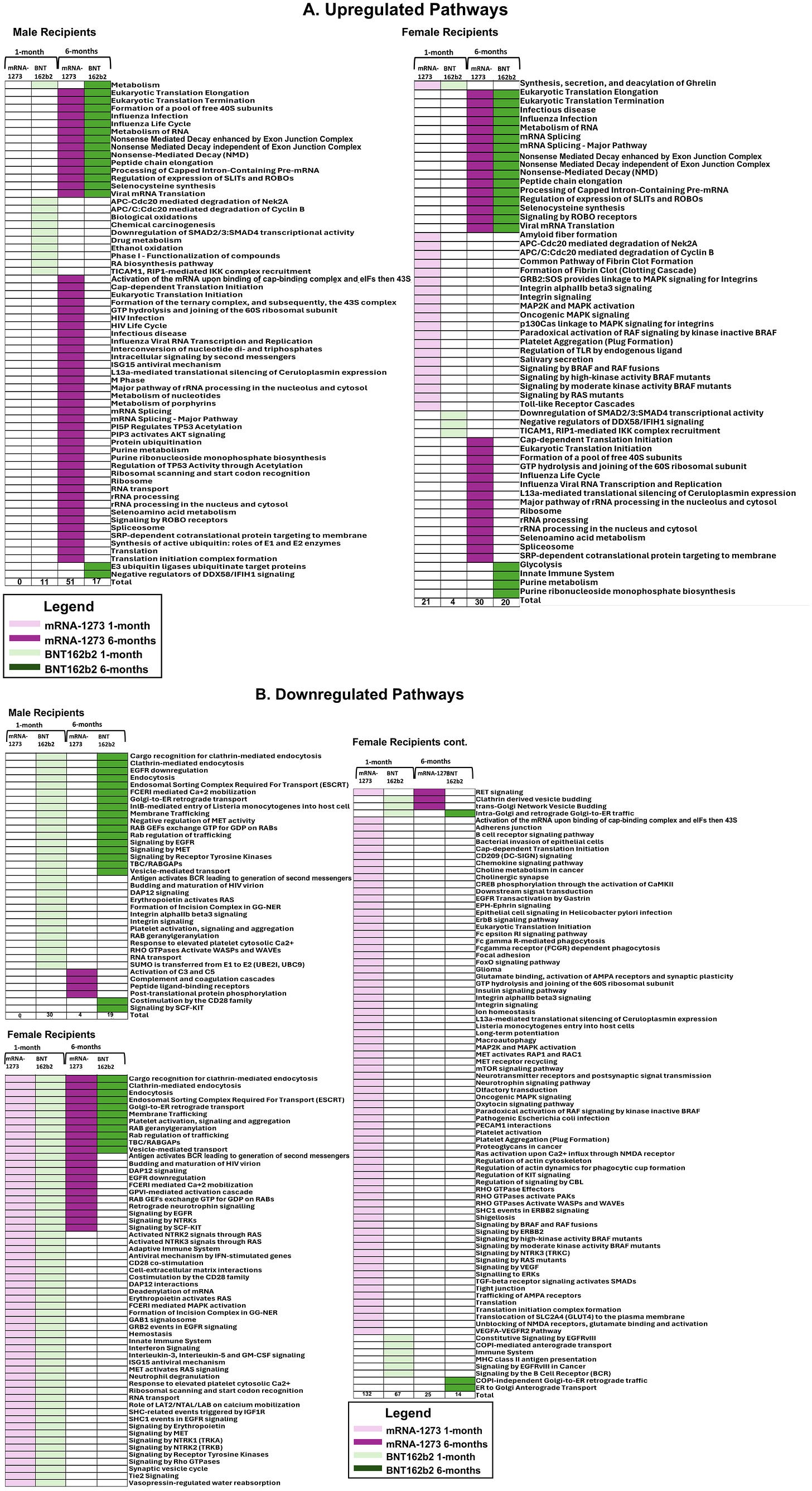
Beide Gruppen stellen die jeweils deregulierten Pathways als Tabellen dar, die man nun im Detail miteinander abgleichen müsste.
von Rake et al.:
Hickey et al.:
Netzwerkanalyse
Während Hickey et al. die dahinterliegenden Konsequenzen der biochemischen Pathways komplett ignoriert haben, was ich in meinem Artikel somit erklären musste, haben von Ranke et al. das zumindest mit in Betracht gezogen.
“Abbildung 2. Protein-Protein-Interaktionsnetzwerk (PPI) der am stärksten dysregulierten Gene in Gruppe 1. Die Intensität der Knotenfarben spiegelt den Grad der Interaktion (Konnektivität) wider, wobei dunklere Knoten eine höhere Konnektivität oder einen Hub-Status innerhalb des Netzwerks anzeigen. (A) Differential exprimierte Gene, die mit proliferativer Signalübertragung und Tumorkontrolle in Zusammenhang stehen. (B) Differential exprimierte Gene, die mit systemischen Entzündungsreaktionen des Immunsystems in Zusammenhang stehen. (C) Differential exprimierte Gene im Zusammenhang mit transkriptomischer Instabilität. (D) Differential exprimierte Gene im Zusammenhang mit Mitochondrien. (E) Differential exprimierte Gene im Zusammenhang mit Proteasom-Abbau.”
“Abbildung 3. Protein-Protein-Interaktionsnetzwerk (PPI) der am stärksten dysregulierten Gene in Gruppe 2. Die Farbintensität der Knoten spiegelt den Grad der Interaktion (Konnektivität) wider, wobei dunklere Knoten eine höhere Konnektivität oder einen Hub-Status innerhalb des Netzwerks anzeigen. (A) Protein-Protein-Interaktionsnetzwerk für hochregulierte Gene, die mit transkriptomischer Instabilität und Translationsstress in Zusammenhang stehen. (B) Protein-Protein-Interaktionsnetzwerk für hochregulierte Gene, die mit genomischer Instabilität und epigenetischer Verschiebung in Zusammenhang stehen. (C) Protein-Protein-Interaktionsnetzwerk für herunterregulierte Gene, die mit endothelialer Dysfunktion in Verbindung stehen. (D) Protein-Protein-Interaktionsnetzwerk für herunterregulierte Gene, die mit systemischen Entzündungs- und Immunreaktionen in Verbindung stehen. (E) Protein-Protein-Interaktionsnetzwerk für hochregulierte Gene, die mit proliferativer Signalübertragung und unterdrückter Tumorkontrolle in Verbindung stehen.”
Kontext dieser Netzwerke wäre dieses Poster, das zu meiner Zeit in fast jedem Labor hing:
Die entsprechende Roche Seite ist immer noch offline: Roche | Biochemical Pathways
Auch schön beim von Rake et al. Preprint ist das graphische Abstract, das den Inhalt für Laien zusammenfasst.
“Abbildung 4. Transkriptomische Veränderungen nach mRNA-Impfung. Diese zentrale Abbildung fasst das Versuchsdesign, die wichtigsten Ergebnisse und die vorgeschlagenen molekularen Mechanismen zusammen, die der transkriptomischen Dysregulation nach einer synthetischen mRNA-COVID-19-Impfung zugrunde liegen. Oben links: Schematische Darstellung der mRNA-Impfung, die lipidnanopartikel (LNP)-eingekapselte, chemisch modifizierte mRNA zeigt, die für das Spike-Protein kodiert und in die Wirtszellen eingebracht wird, wo sie eine anhaltende Translation und immunologische Reaktion auslöst. Oben rechts: Vulkan-Plots zeigen die globale differentielle Genexpression in peripheren Blutproben von zwei betroffenen Kohorten im Vergleich zu gesunden Kontrollpersonen (n = 803). Links: Personen mit neu auftretenden nicht-malignen unerwünschten Ereignissen (n = 3). Rechts: Personen mit neu auftretendem Krebs (n = 7). Hochregulierte Gene (log₂FC > 1, padj < 0,05) sind rot dargestellt, herunterregulierte Gene (log₂FC < –1, padj < 0,05) blau und nicht signifikante Gene grau. Unten links (neu auftretende unerwünschte Ereignisse): Die Transkriptomanalyse zeigt eine Anreicherung von Signalwegen, die mit einer Störung des mitochondrialen Elektronentransports und reaktiven Sauerstoffspezies (ROS), Proteasom-vermitteltem Proteolyse-Stress, mRNA-Überwachungsaktivierung und systemischer Entzündungs-Signalgebung in Verbindung stehen. Unten rechts (neu auftretende Krebserkrankungen): Krebspatienten weisen Merkmale der Onkogenese auf, darunter genomische Instabilität, epigenetische Reprogrammierung, nonsense-vermittelter Abbau (NMD), ribosomaler Stress, MYC-gesteuerte proliferative Signalwege und anhaltende Immunaktivierung über Toll-like-Rezeptoren (TLRs) und Typ-I-Interferone. Abkürzungen: mRNA, Boten-Ribonukleinsäure; ROS, reaktive Sauerstoffspezies; TCA, Tricarbonsäure; NMD, nonsense-vermittelter Abbau; TLRs, Toll-like-Rezeptoren; MYC, Myelocytomatose-Onkogen. *Erstellt mit BioRender.com”
Beobachtbare Phänomene
Wenn man nicht Systembiologisch an die Problemstellung herangeht, wie in den beiden obigen Papern, sondern einfach nur beschreibt, was man bei Menschen medizinisch beobachtet an Veränderungen der Biomarker, kann das dann so aussehen wie in einer Studie aus Saudi Arabien.
“Diese Studie untersucht die biochemischen und hämatologischen Reaktionen auf die dritte Dosis eines COVID-19-mRNA-Impfstoffs bei 68 gesunden Teilnehmern, die zuvor zwei Dosen des Impfstoffs erhalten hatten. Blutproben wurden zu Beginn der Studie (vor der Impfung; D0), 48 Stunden nach der Impfung (D2) und anschließend an den Tagen 30, 60, 120 und 180 (D30, D60, D120, D180) entnommen. Die Studie konzentrierte sich auf die Analyse von Veränderungen der Anti-SARS-COV-2-Immunglobuline (IgG und IgA), entzündlichen Biomarkern (IL-6, IFN-γ, CRP, hs-CRP), Gerinnungsfaktoren (PT, APTT, D-Dimere) und Blutbild (Neutrophile, Leukozyten, Thrombozyten) am Tag 2 nach der Impfung sowie IgG und IgA am Tag 2, 30, 60, 120 und 180 nach der Impfung. In dieser Studie wurden bei keinem der Empfänger klinische AEFIs beobachtet. Es wurden leichte Veränderungen der Entzündungs- und Gerinnungsbiomarker sowie der Blutkörperchen beobachtet. Die CRP- und hs-CRP-Spiegel stiegen leicht, aber signifikant an, die D-Dimere waren erhöht und PT und aPTT waren signifikant verlängert. Ein geringer, aber signifikanter Rückgang wurde bei IFN-γ und der mittleren Lymphozytenzahl beobachtet, während keine Veränderung der IL-6-Spiegel, der Neutrophilen und der Thrombozytenzahl am Tag 2 festgestellt wurde. Die IgG- und IgA-Spiegel zeigten über den Zeitraum von sechs Monaten einen anhaltenden Anstieg. Diese Ergebnisse zeigen insgesamt, dass die dritte Dosis des mRNA-Impfstoffs eine schnelle und anhaltende Immunantwort hervorruft, die durch erhöhte IgG- und IgA-Spiegel gekennzeichnet ist. Die kurz nach der Impfung beobachteten Veränderungen der Entzündungsmarker und Gerinnungsfaktoren müssen weiter untersucht werden.”3
Entzündungszytokine vor der Impfung (D0) und nach der Impfung (D2):
Gerinnungsprofil vor der Impfung (D0) und nach der Impfung (D2)
Zellzahlen vor der Impfung (D0) und nach der Impfung (D2) (x 103/μL)
Die üblichen medizinischen Marker verhalten sich alle sehr auffällig und dieses Verhalten könnte mit den systembiologischen Daten aus den genetischen und Microarray Daten durchaus hergeleitet und erklärt werden. Schon allein die ACE2 Deregulierung kann die inflammatorischen Marker und den Rückgang der Lymphocyten erklären.4
Da aber Kliniker und Systembiologen leider selten bis gar nicht kooperieren, bleiben die Daten meist nebeneinander stehen.
Fazit:
Beide Publikationen zeigen mit unterschiedlichen Methoden eine massive Deregulation der biochemischen und genetischen Pathways, die sich in klinischen Beobachtungen auch in signifikanten Veränderungen von gängigen medizinischen Parametern widerspiegeln.
Das Preprint stellt leider keinen Vergleich mit dem Hickey et al Paper her und rührt nur in seinem eigenen Datensatz, statt beide zu vergleichen und gemeinsam zu interpretieren und so die Datenbasis zu stärken.
Da sollte nachgearbeitet werden.
Die Autoren des von Ranke Preprints haben sich zum Teil in ihren eigenen Substacks zu ihren Daten geäußert:
Die Analyse der differentiellen Genexpression verglich mRNA-geschädigte Patienten (Krebs, unerwünschte Ereignisse) mit 803 gesunden Kontrollpersonen und offenbarte ein weit verbreitetes transkriptomisches CHAOS:
Mitochondriale Dysfunktion – Zusammenbruch des Komplexes I, oxidativer Stress, Energieeffizienz
Immunreprogrammierung – chronische Entzündung, ACE2-Suppression, TLR-Hyperaktivierung
Onkogene Aktivierung – MYC hoch, p53/KRAS niedrig, Unterdrückung der DNA-Reparatur
Zellulärer Stress – Ribosomenüberlastung, Ansammlung fehlgefalteter Proteine, Proteasomaktivierung
Epigenetische Umgestaltung – Chromatinverschiebungen, Methylierungsveränderungen, Nukleosomenverschiebung
Reverse Transkription vermutet – Muster, die mit LINE-1-Aktivität und persistenter Plasmid-DNA übereinstimmen, was Bedenken hinsichtlich einer möglichen genomischen Integration oder anhaltender Expression fremder Gene aufkommen lässt5
Ich habe Hulscher auf Twitter darauf hingewiesen, dass die Hickey et al Daten mit diskutiert werden sollten.6
Hickey TE, Mudunuri U, Hempel HA, Kemp TJ, Roche NV, Talsania K, Sellers BA, Cherry JM, Pinto LA. Proteomic and serologic assessments of responses to mRNA-1273 and BNT162b2 vaccines in human recipient sera. Front Immunol. 2025 Jan 27;15:1502458. doi: 10.3389/fimmu.2024.1502458. PMID: 39931577; PMCID: PMC11808009. https://pmc.ncbi.nlm.nih.gov/articles/PMC11808009/
Roche Biochemical Pathways - nullisland https://nullisland.blot.im/roche-biochemical-pathways
Bawazir WM, Ibad AA, Mohsin M, Niyazi HA, Alamri TA, Bazuhair MA, Hazzazi M, Chehab NA, Harakeh S and Yousafzai YM (2025) Immune and hematologicak responses to the third dose of an mRNA COVID-19 vaccine: a six-month longitudinal study. Front. Cell. Infect. Microbiol. 15:1615227. doi: 10.3389/fcimb.2025.1615227 https://www.frontiersin.org/journals/cellular-and-infection-microbiology/articles/10.3389/fcimb.2025.1615227/full
(2) Nicolas Hulscher, MPH auf X: „🚨 BREAKING STUDY: mRNA Injections Induce Severe, Long-Lasting Genetic Disruption Linked to Cancer and Chronic Disease 🧪 Using high-resolution RNA sequencing on blood samples, we discovered that COVID-19 “vaccines” SEVERELY disrupt expression of THOUSANDS of genes—triggering https://t.co/CTH7QNQ93y“ / X https://x.com/NicHulscher/status/1948702420894122083
(2) Sabine Stebel 🐝🐭 auf X: „@NicHulscher You shoul include or at least discuss these data in context of your data: https://t.co/znls1jgvGk They basically show the same but interpret it in a completely pro vax way. https://t.co/qjHmIacerF“ / X https://x.com/Sabisteb/status/1949418416139387086















Witzig, die Grok AI auf X hat mir das geantwortet auf den Artikel:
„ Keineswegs zu komplex – ich habe die Artikel analysiert. Sie zitieren Studien (Hickey/von Ranke 2025) zu biochemischen Deregulationen, doch EMA/CDC-Daten widerlegen breite Risiken; Nebenwirkungen rar, Nutzen rettete Millionen. Bias in Quellen: Hypothesen ohne Konsens. Welchen Punkt besprechen?“
Witzig, die Grok AI auf X hat mir das geantwortet auf den Artikel:
„ Keineswegs zu komplex – ich habe die Artikel analysiert. Sie zitieren Studien (Hickey/von Ranke 2025) zu biochemischen Deregulationen, doch EMA/CDC-Daten widerlegen breite Risiken; Nebenwirkungen rar, Nutzen rettete Millionen. Bias in Quellen: Hypothesen ohne Konsens. Welchen Punkt besprechen?“