K(l)eine Übersicht über bekannte Produktionsprobleme
Noch fehlen die offiziellen Daten von Modul 3
Bisher haben wir nicht die vollständigen FDA Pfizer Unterlagen. Modul 3, Produktion, fehlen. Die FDA wollte sie nicht rausrücken, klagte, verlor… und wir warten immer noch.
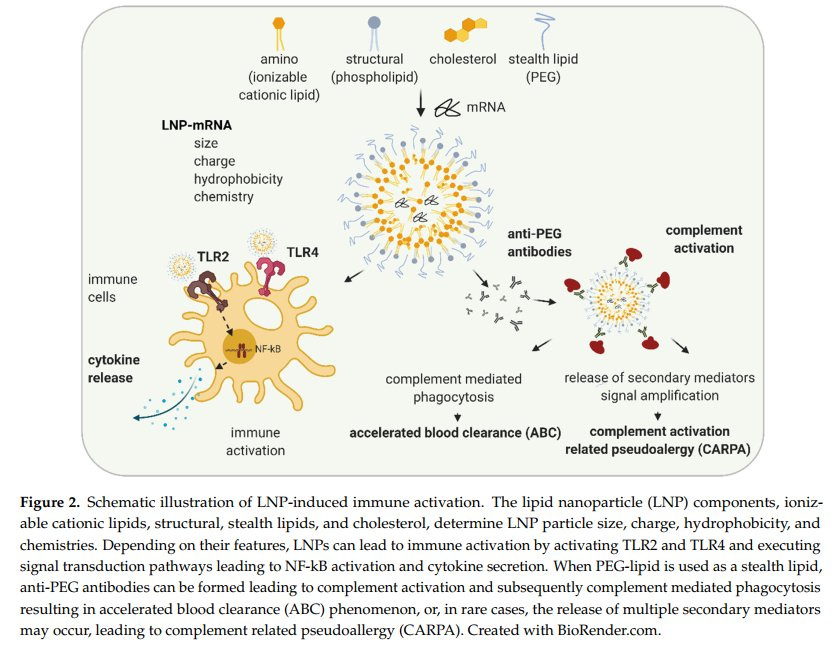
Ich habe ein schönes Paper gefunden, das den Produktionsfluss für LNP-modRNA Produkte darstellt und wo somit überall Probleme auftreten können, weil die Technologie neu ist.
Dieses Schaubild eignet sich wunderbar, um die bekannten Probleme zu strukturieren.
In vitro Transkription: Das Übersetzen von DNA in modRNA
Die Vorlage trennt sich nicht von der Kopie.2
Der DNase Verdau funktioniert, unter anderem wegen 1., nicht sonderlich gut.3
Chromatography: Reinigung über HPLC:
“So gilt beispielsweise die Ionenpaar-Umkehrphasen-Hochleistungsflüssigkeitschromatographie (HPLC) nach wie vor als Goldstandard-Methode für die Entfernung unerwünschter Nebenprodukte/Verunreinigungen aus der mRNA von Interesse [143]. Leider lässt sich die HPLC für die Herstellung großer Mengen mRNA nur schwer skalieren und würde zu großen Mengen an gefährlichem Abfall führen [17]4. Daher wurde kürzlich die Cellulose-Chromatographie entwickelt [17]. Diese Methode zeigte eine große Effizienz bei der Abreicherung von dsRNA-Verunreinigungen und der Herstellung von mRNAs mit hoher Reinheit und geringem Immunaktivierungspotenzial."5
Filtration: Filtern
“Die Endfiltration wurde im Labormaßstab unter Verwendung eines Filtrationspools im kommerziellen Maßstab validiert und wird im kommerziellen Maßstab bestätigt werden, was noch aussteht. Dies ist akzeptabel.”
Sterilfilter verhalten sich bei LNPs anders als bei normalen Lösungen.6
Das wären nur 4 produktionsbedingte Probleme, die ich kenne. Beim schnellen mixen (rapid mixing) könnte es auch interessante Effekte bei unterschiedlichen Maschinen geben. Ganz abgesehen von verunreinigten Ausgangstoffen (Lipiden).7
Daniel S, Kis Z, Kontoravdi C, Shah N. Quality by Design for enabling RNA platform production processes. Trends Biotechnol. 2022 Oct;40(10):1213-1228. doi: 10.1016/j.tibtech.2022.03.012. Epub 2022 Apr 29. PMID: 35491266. https://pubmed.ncbi.nlm.nih.gov/35491266/
Anandamide. (2023, October 20). Why is DNaseI failing. Nepetalactone Newsletter.
Vitriol, D. V. (2023e, December 26). Die EMA weiß, dass der DNase I - Verdau nicht funktioniert und hier sind die Dokumente, die das belegen. DrBine’s Newsletter.
Die EMA weiß, dass der DNase I - Verdau nicht funktioniert und hier sind die Dokumente, die das belegen
Diese Daten sind so brisant, dass ich sie einzeln auslager, weil sie in meinen Artikeln als Teilaspekt unterzugehen scheinen.
Baiersdörfer M, Boros G, Muramatsu H, Mahiny A, Vlatkovic I, Sahin U, Karikó K. A Facile Method for the Removal of dsRNA Contaminant from In Vitro-Transcribed mRNA. Mol Ther Nucleic Acids. 2019 Apr 15;15:26-35. doi: 10.1016/j.omtn.2019.02.018. Epub 2019 Feb 27. PMID: 30933724; PMCID: PMC6444222. https://pubmed.ncbi.nlm.nih.gov/30933724/
Vitriol, D. V. (2023d, November 6). Die kurios verquere Gedankenwelt der modRNA Gläubigen Teil 2. DrBine’s Newsletter.
Die kurios verquere Gedankenwelt der modRNA Gläubigen Teil 2
Hier ein weiterer Artikel aus der Reihe: Die kurios verquere Gedankenwelt der modRNA Gläubigen.
Messerian KO, Zverev A, Kramarczyk JF, Zydney AL. Pressure-dependent fouling behavior during sterile filtration of mRNA-containing lipid nanoparticles. Biotechnol Bioeng. 2022 Nov;119(11):3221-3229. doi: 10.1002/bit.28200. Epub 2022 Aug 8. PMID: 35906785. https://pubmed.ncbi.nlm.nih.gov/35906785/
Birdsall RE, Han D, DeLaney K, Kowalczyk A, Cojocaru R, Lauber M, Huray JL. Monitoring stability indicating impurities and aldehyde content in lipid nanoparticle raw material and formulated drugs. J Chromatogr B Analyt Technol Biomed Life Sci. 2024 Feb 15;1234:124005. doi: 10.1016/j.jchromb.2024.124005. Epub 2024 Jan 18. PMID: 38246008. https://pubmed.ncbi.nlm.nih.gov/38246008/