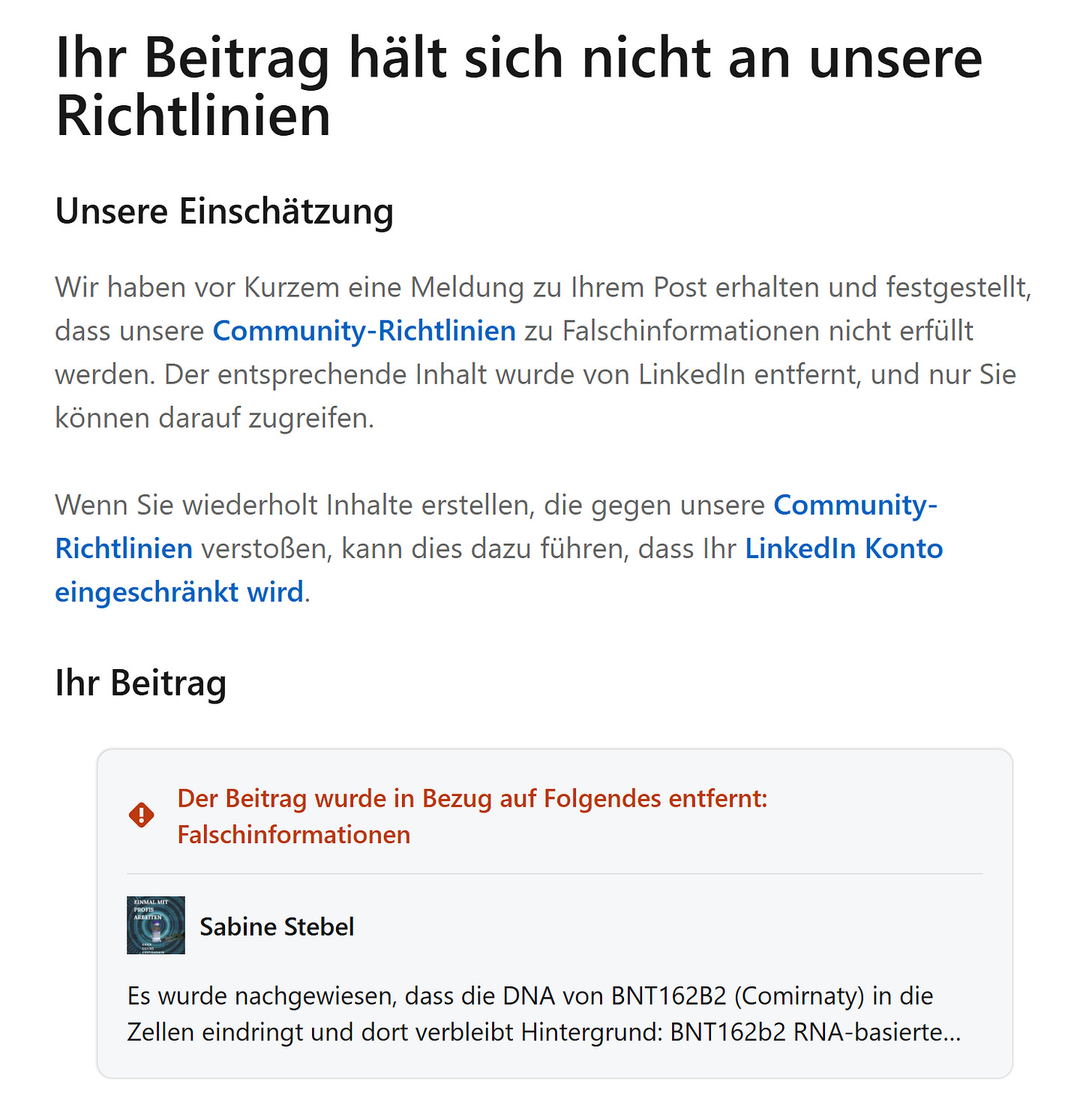
Es wurde nachgewiesen, dass die DNA von BNT162B2 (Comirnaty) in die Zellen eindringt und dort verbleibt
BioNTech RNA-Based COVID-19 Injections Contain Large Amounts Of Residual DNA Including An SV40 Promoter/Enhancer Sequence
Frisch aus der Druckerpresse:
Kämmerer U, Schulz V, Steger K. BioNTech RNA-Based COVID-19 Injections Contain Large Amounts Of Residual DNA Including An SV40 Promoter/Enhancer Sequence. Science, Public Health Policy and the Law. 2024 Dec 03; v5.2019-2024 https://publichealthpolicyjournal.com/biontech-rna-based-covid-19-injections-contain-large-amounts-of-residual-dna-including-an-sv40-promoter-enhancer-sequence/
Wie üblich gilt, lest das komplette Paper, das ist nur ein stark gekürzter Werbeblock.
Also die deutsche Übersetzung der spannenden Teile für all jene, die kein Englisch können.
Hier die deutsche Übersetzung der Highlights.
Hintergrund: BNT162b2 RNA-basierte COVID-19-Injektionen dienen der Transfektion menschlicher Zellen zur effizienten Produktion von Spike-Proteinen für eine Immunreaktion.
Ergebnisse: Wir konnten eine erfolgreiche Transfektion von nukleosidmodifizierter mRNA (modRNA) in HEK293-Zellen nachweisen und zeigen robuste Konzentrationen von Spike-Proteinen über mehrere Tage der Zellkultur. Die Sekretion in den Zellüberstand erfolgte überwiegend über extrazelluläre Vesikel, die mit Exosomenmarkern angereichert sind. Wir haben den RNA- und DNA-Gehalt dieser Fläschchen weiter analysiert und nach dem RNase-A-Verdau in allen Chargen große DNA-Mengen mit Konzentrationen zwischen 32,7 ng und 43,4 ng pro klinischer Dosis festgestellt. Dies übersteigt bei weitem die maximal zulässige Konzentration von 10 ng pro klinischer Dosis, die von internationalen Aufsichtsbehörden festgelegt wurde. Genanalysen mit ausgewählten PCR-Primerpaaren zeigten, dass die Rest-DNA nicht nur Fragmente der DNA-Matrizen darstellt, die für das Spike-Gen kodieren, sondern von allen Genen des Plasmids, einschließlich des SV40-Promotors/Enhancers und des Antibiotikaresistenz-Gens.
Schlussfolgerung: Unsere Ergebnisse geben Anlass zu ernsten Bedenken hinsichtlich der Sicherheit des BNT162b2-Impfstoffs und fordern einen sofortigen Stopp aller RNA-Biologika, solange diese Bedenken nicht ausgeräumt werden können.
Spike-Protein-Expression in HEK293-Zellen nach Transfektion mit BNT162b2-Biologicals. A: Färbung von Spike-Proteinen in Zellen, die mit verschiedenen Chargen transfiziert wurden, dargestellt in grüner Farbe durch Immunhistochemie. Nicht-transfizierte Zellen (Kontrolle) zeigen keine Färbung. Hämatoxylin dient als Gegenfärbung der Zellkerne.
B: Hellfeldmikroskopie von transfizierten HEK293-Zellen mit verschiedenen Chargen zeigt eine Anhäufung von intrazellulärer Vesikelbildung.
C: Quantifizierung der intrazellulären Spike-Protein (SP)-Konzentrationen über die Zeit, gemessen mittels ELISA. n=2; Mittelwert+SEM.
D: Quantifizierung der sekretierten Spike-Protein (SP)-Konzentrationen im Laufe der Zeit, gemessen mit ELISA. Bei der Charge HD9869 lag die Konzentration des sekretierten Spike-Proteins unter der Nachweisgrenze. n=2; Mittelwert+SEM. Die Negativkontrolle zeigte keinen Spike-Protein-Gehalt im Zelllysat und Zellüberstand. SEM, Standardfehler des Mittelwerts.
DNA-Gehalte in den angegebenen Fläschchen von BioNTech-Chargen vor der RNase A-Behandlung, ohne und mit Triton-X-100-Behandlung. Der Faktor steht für den Anstieg der DNA bei der Freisetzung von Nukleinsäuren aus dem durch Triton gebrochenen LNP. SEM, Standardfehler des Mittelwerts.
CRNA- und DNA-Gehalte (nach Triton-X-100-Behandlung) in den angegebenen Fläschchen der BioNTech-Lose vor und nach der RNase A-Behandlung. Der Faktor stellt den Multiplikator für die Überschreitung des Grenzwerts gemäß der EMA-Verordnung von 0,33ug dsDNA pro 1mg RNA dar. SEM, Standardfehler des Mittelwerts.
Die Polymerase-Kettenreaktion (PCR) zeigt DNA-Reste des kompletten Plasmids, das für die modRNA-Produktion verwendet wurde.
A: Schematische Darstellung des von BioNTech als Vorlage für die modRNA-Produktion verwendeten DNA-Plasmids. Spezifische Sequenzen (SV40-Promotor/Enhancer; Neomycin-resistente Kassette, ORI-Replikon, Spike-Protein) sind farblich hervorgehoben, ebenso wie die zum Nachweis dieser Gene verwendeten Sequenzen, die von 1 bis 7 nummeriert sind.
B: Visualisierte DNA-Fragmente nach PCR der angegebenen Sequenzen (Abbildung A, 1-7) zeigen den Nachweis aller Gene aus dem DNA-Plasmid in den Fläschchen (rechts, 1:10 verdünnt vor dem Laden zur PCR) und in Plasmidpräparationen aus Zellen, die mit diesen biologischen Stoffen transfiziert wurden (links). Mock steht für nicht-transfizierte Zellen als Negativkontrolle für den linken Teil und Wasser als Negativkontrolle für den rechten Teil. Die Partie GH9715 diente als Positivkontrolle.
Die Polymerase-Kettenreaktion (PCR) zeigt DNA-Reste des kompletten Plasmids, das für die modRNA-Produktion verwendet wurde.
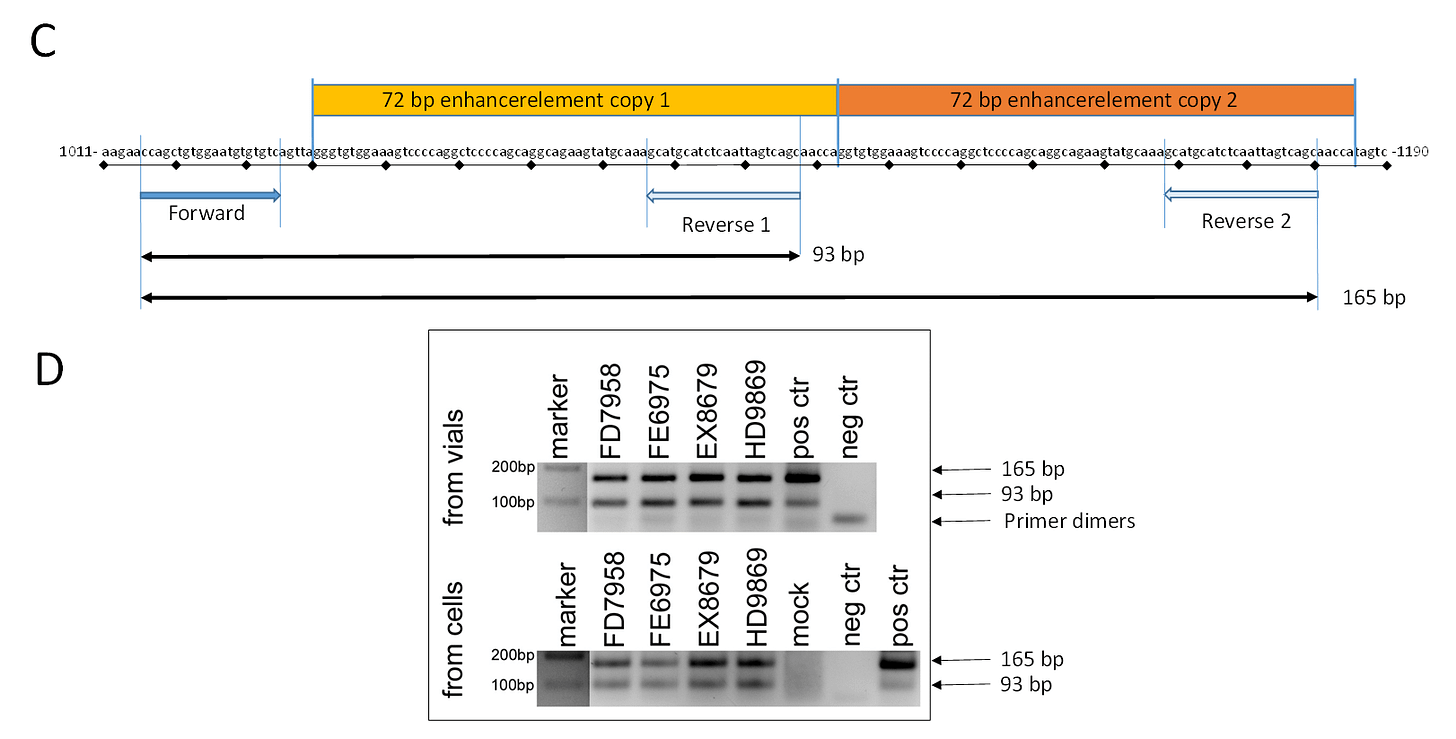
C: Die Karte der SV40-Promotor/Enhancer-Region zeigt die beiden Kopien des 72bp-Elements, die zu den beiden Amplikons führen, die in der PCR aufgrund der beiden Bindungsstellen des Reverse-Primers in Kombination mit der einzigen Bindungsstelle des Forward-Primers sichtbar sind.
D: Zusätzliche PCR für den SV40-Promotor/Enhancer (gleiche PCR-Bedingungen wie in Abbildung 3B mit Ausnahme einer längeren Laufzeit), um die beiden Amplikons bei 165 bp und 93 bp besser zu trennen und sichtbar zu machen.
Schlussfolgerung
Wir konnten zeigen, dass die Transfektion der menschlichen Zelllinie HEK293 mit vier verschiedenen BNT162b2-Chargen über mehrere Tage zur Produktion von Spike-Proteinen führt, die über Exosomen in den Zellüberstand abgegeben werden. Wir wiesen in allen Fläschchen Restplasmid-DNA in Konzentrationen nach, die weit über dem zulässigen EMA-Grenzwert von 0,33 ng dsDNA pro 1 mg RNA lagen. Wir identifizierten alle Plasmid-Gene sowie die beiden Kopien des SV40-Promoter/Enhancer-Elements. Es wurde nachgewiesen, dass die DNA in die Zellen gelangt und dort verbleibt.
Kleiner Hinweis zu Exosomen und Shedding. Die Spikes sollten eigentlich in der Membran bleiben ABER, da das Spike-Protein im Endoplasmatischen Retikulum produziert wird, war mit Mittelstufenwissen klar, dass es Exosomen geben muss.
Mögliche biochemische Wege zu shedden
Eigentlich sind die Puzzlestücke schon alle irgendwo in meinen Artikeln, aber ich fasse sieh hier einmal in einem gemeinsamen Artikel zusammen und strukturiere sie um. Text- und Buchstabenrecycling, um die Umwelt zu schonen. Wer weiß, vielleicht werden in Deutschland irgendwann sogar dich Buchstaben knapp. Darf ich das „Z“ mittlerweile wieder verwenden …
[…]
Unsere Ergebnisse bestätigen und erweitern veröffentlichte Berichte und geben Anlass zu ernsten Bedenken hinsichtlich der Sicherheit des BNT162b2-Impfstoffs. Wir fordern einen sofortigen Stopp aller biologischen Impfstoffe auf RNA-Basis, bis diese Bedenken wissenschaftlich geklärt und überzeugend ausgeräumt sind.
Weitere Analysen des Papers:
Das Paper verstößt im übrigen gegen die LinkedIn Richtlinien:
Der Grund, warum dieser Post wohl auf dem Schirm der Zensoren landete, im Gegensatz zu meinen anderen, könnte dieser sein:
Erklärbär Video von Ulrike Kämmerer zum Paper:













As I have been saying for 2.5 yrs regarding the exosomes!. Similar in mechanism of action of Onpattro. The cell gets rid of the excess spike via exosomes which can travel "all over the body" and can also transfect. It is NOT the LNPs which goes throughout the body, they target the organs with fenestrated epithelium.
What I want to know is if there is also mRNA in those exosomes, and broken down LNPs.
Eventuell von Interesse. Mir fehlen leider die Kenntnisse um aus der Studie evtl. wichtige Schlussfolgerungen abzuleiten.
„Exosomal vaccines containing the S protein of the SARS coronavirus induce high levels of neutralizing antibodies“/2007
...Wir untersuchten daher Impfstoffe auf Exosomenbasis, die die Spike-S-Proteine von SARS-CoV enthalten. S-haltige Exosome wurden durch Ersetzen der transmembranen und zytoplasmatischen Domänen des S-Proteins durch die von VSV-G gewonnen. Die Immunogenität und Wirksamkeit der S-enthaltenden Exosomen wurde an Mäusen getestet und mit einem Adenovirus-Vektor-Impfstoff verglichen, der das S-Protein exprimiert...
https://pmc.ncbi.nlm.nih.gov/articles/PMC7103344/