Ugurs grenzdebile Schwachsinnsideen im Protein Design
Teil 5: Welche (biochemischen) Basisdaten des Spikes ich erhoben hätte = meine Wunschliste an BioTNech/Pfizer
Nachdem wir nun geklärt haben, welche Anfängerfehler von Ugur und seiner Crew gemacht wurden, ist es an der Zeit zu beschreiben, welche Analysen ich durchgeführt hätte, hätte ich noch Zugang zu meinem alten Labor und der entsprechenden Ausrüstung. Schön wäre es, wenn BioNTech/Pfizer diese Daten nachliefern würden und zwar als Rohdaten ohne digitale Bearbeitung, damit unabhängige Wissenschaftler diese fachlich überprüfen können.
JETZT wird es speziell. Das ist mein Wunschzettel an Analysen, die ich bei den Pfizer Daten bisher schmerzlich vermisse. Dieser Artikel richtet sich eher an jene, die ein Labor haben und verstehen, was ich hier gerne hätte. Der normale Leser braucht sich daher nicht zu wundern, wenn er nur noch Bahnhof versteht. Macht euch keine Sorgen, BioNTech/Pfizer haben es auch nicht verstanden.
Es geht mir daher nicht darum, dass der gelegentliche Leser, der sich in diesem fünften Teil verirrt, im Detail versteht, was nicht gemacht wurde. Es geht darum grob zu verstehen, dass grundlegende, teils sehr, sehr alte, etablierte Methoden der Charakterisierung des Produkte NICHT durchgeführt wurden und was alles bisher unbekannt ist.
Man könnte das auch als meinen persönlichen Fragekatalog an BioNTech/Pfizer ansehen. Oder als meinen Weihnachtswunschzettel an mitlesende Wissenschaftler.
Charakterisierung der modRNA
Fragen, die sich mir bezüglich der modRNA stellen und zu denen ich bisher KEINE DATEN gefunden habe wären:
1. Wieviel % der Uracile wurden durch N1-Methylpseudouridin[1] ersetzt? 100%? 90%? Hat man das bestimmt? Wenn ja, wie hat man das bestimmt?
2. Kann man die modRNA mit verschiedenen menschlichen RNAsen verdauen? Wie sieht das im zeitlichen Verlauf aus? Z. Bsp. nach 5s, 10s, 30s, 60s? Bitte einmal mit Lipidhülle intakt und einmal ohne Lipidhülle z. Bsp. nach Isoprop:Ethanol Fällung.
3. Sind miRNA (microRNA) Fragmente enthalten, welche regulatorische Funktion ausüben könnten?
4. Sind in den Produkten Oncomir Sequenzen enthalten?
5. Wie groß ist die Wahrscheinlichkeit, dass die Stop-Codons wegen des verwendeten durch N1-Methylpseudouridin durchgelesen werden oder Rasterschübe passieren?
Integriert die transkribierte modRNA oder die Plasmid-Verunreinigung in den Zellkern?
Mittlerweile ist bekannt, dass das Impfprodukt in den Zellkern integrieren kann.
BioNTech/Pfizer selbst wussten zumindest, dass das Spike-Protein in den Zellkern geht. Das erkennt man am sogenannten FRET-Signal, weil grün + blau andere Farben ergibt, wegen Energieübertragung zischen den Farbstoffen.
Nicht bekannt ist, was integriert. Die (mittels reverser Transkriptase LINE-1) in DNA übersetzte modRNA oder das als Verunreinigung identifizierte Plasmid[3], welches in der Produktion als Vorlage für die modRNA diente?
Im Prinzip führt man mit diesen Schlumpfungen ein altes, bewährtes Experiment durch, welches man in der Zeit vor der Sequenzierung verwendet hat, um Krebsgene (Oncogene) oder Tumorsuppressorgene zu identifizieren. Man nutzte sogenannte springende Gene (Transposons) oder Retroviren und ließ diese zufällig irgendwo integrieren. Anschließend beobachtete man, ob die Zelle entartete und schaute, welches Gen dadurch zerstört wurde[4].
Mit dieser Methodik könnte man auch herausfinden, was tatsächlich integriert hat und Spike produziert. Wenn man einige der sogenannten „Turbokrebse“ daraufhin untersuchen würde, welches Gen entartet ist, und warum, könnte man identifizieren, was (Plasmid oder transkribierte modRNA) wo integriert hat. Kann man die transkribierte modRNA nachweisen? Kann man modRNA nachweisen? Kann man das Plasmid nachweisen?
Das Problem der potentiellen Integration des „Impfstoffes“ in das Genom ist durchaus bekannt.[5]
Das wäre einfach mit Northern[6] (RNA-Nachweis) und Southern Blots[7] (DNA-Nachweis) möglich.
Aufgrund der Plasmidverunreinigung ist es aktuell unklar, ob das von Pathologen nachgewiesene Spike Protein tatsächlich von der stark modifizierten modRNA hergestellt wird, oder es vielleicht immer die Plasmide waren. Da die erste Schlumpfung auf Plasmidbasis (doppelsträngige DNA) mittlerweile zugelassen wurde, wäre es wichtig zu wissen, ob das modRNA Plasmid überhaupt jemals funktioniert hat oder es immer eine Plasmid-Impfung war wie das indische ZyCoV-D[8]. Damit wäre potentiell die komplette modRNA Plattform in Frage gestellt.
Grundlegende Proteincharakterisierung
Exprimieren Blutzellen das Spike Protein
Man könnte recht einfach mittels FACS[9] herausfinden, ob Blutzellen Geimpfter (welcher Art auch immer) das Spike-Protein an ihrer Oberfläche präsentieren.
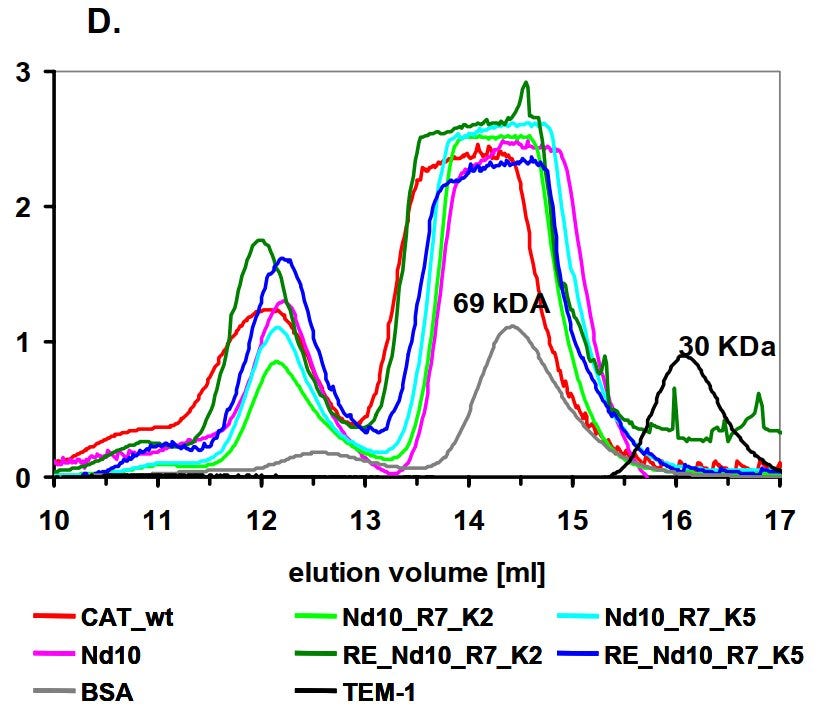
Bildet das Spike Multimere?
Meiner eigenen Laborerfahrung nach können Proteine, die aus mehreren Untereinheiten bestehen, verschiedene Varianten in einem dynamischen Gleichgewicht ausbilden. Bei einem Trimeren Protein (also ein Protein aus drei Untereinheiten) theoretisch in einem Monomer-Dimer-Trimer- Hexamer-Nonamer-Gleichgewicht stehen. Das könnte man auf einer Sephadex 200 Säule sehen, wenn dieses Verhältnis entsprechend stabil ist. In meiner Promotion war es ein Trimer-Hexamer-Nonamer Gleichgewicht.
Wie es beim Spike aussieht, ist unbekannt.
Erschwerend kommt hinzu, dass hybride Spikes durch Kreuzimpfung zu erwarten sind, da die Spike-Protein der verschiedenen Hersteller sich minimal unterscheiden.
BioNTech/Pfizer unterscheidet sich von Moderna und Janssen durch die Codonoptimierung, was zu unterschiedlichen Konformationen führen kann.
AstraZenecas Spike hat kein Prolin-Schloss.
Es kann also bei einer Kreuzimpfug von Jannsen, AstraZeneca, BioNTech/Pfizer, BioNTech/Pfizer Bivalent zur Bildung von Trimeren aus minimum vier verschiedenen Untereinheiten kommen. Wie sich entsprechende hybride Spike-Proteine verhalten ist unbekannt. Zusätzlich handelt es sich um ein dynamisches Gleichgewicht mit häufigem Partnerwechsel unter den Untereinheiten, denn alles fließt.
Welche Schmelz- oder Denaturierungstemperaturen hat das Spike?
Proteine können ausflocken/denaturieren. Es gibt Hitzedenaturierung, wie man das vom Rührei kennt, aber es gibt auch Kältedenaturierung, also dass ein Protein ausflockt, weil ihm zu kalt ist.
Es gäbe einfache, low tech Möglichkeiten einen groben Überblick zu bekommen, wie sich das Spike-Protein bei verschiedenen Temperaturen verhält.
Hitzedenaturierung wäre im menschlichen Körper kein Problem, denn die ersten unserer körpereigenen Proteine denaturieren um die ca. 43°C. So ein hohes Fieber kann tödlich enden, weil eben lebenswichtige Protein ausfallen. Wäre das Spike-Protein thermostabil, würde das in dieser Hinsicht kein Problem darstellen.
Das Problem jedoch ist, dass vor allem thermostabile Proteine gerne kältedenaturieren, also bei einstelligen Temperaturen bereits ausflocken. Das war damals in der Promotion eines meiner Probleme. Mein Protein mochte es kuschelig warm von 37°C aufwärts. 4°C hat es ausflocken oder regelrecht fest werden lassen. Man konnte das Reagenzglas umdrehen und es blieb als feste, leicht trübe Substanz drinnen haften.
Es wurde beobachtet, dass Serum geimpfter Personen im Kühlschrank fest werden kann. Das ist der Effekt, den ich bei meinem Protein beobachtet habe und könnte somit auf ein kältedenaturierendes Protein hinweisen. Das wäre zu klären.
Eine einfache Methode hierfür wäre, Spike Protein, welches aus Impfgeschädigten per Blutwäsche entfernt wurde, verschiedenen Temperaturen auszusetzen. Anschließend, abzuzentrifugieren und dann mittels SDS-Gel und Western-Blot zu prüfen ab welcher Temperatur, das Spike Protein im abzentrifugierten Serum nicht mehr nachweisbar ist.
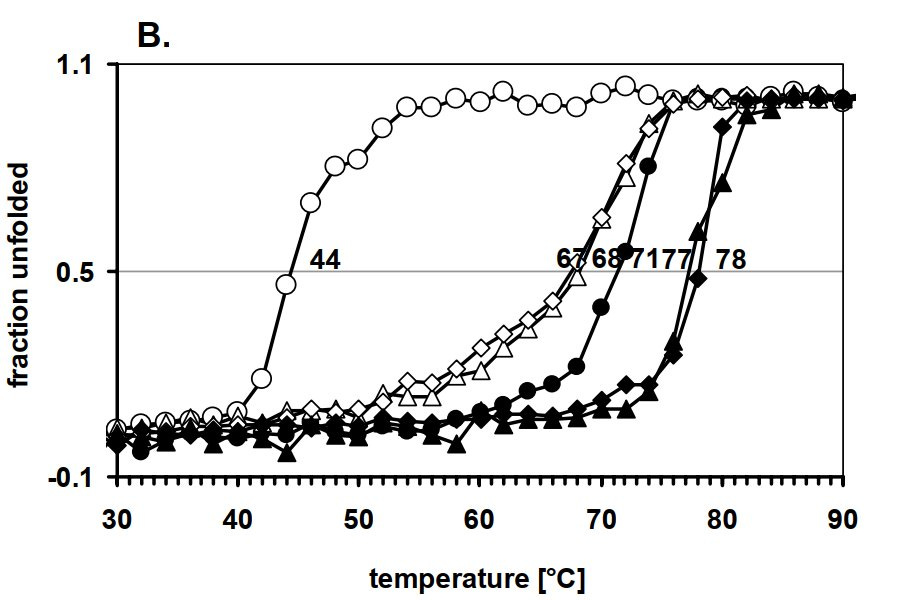
Eleganter ginge es mit einer CD-Schmelzkuve. CD steht für circular dichroism[11] und damit kann man messen, bei welcher Temperatur ein Protein seine Struktur durch Denaturierung verliert.
So eine CD-Schmelzkurve kann wie folgt aussehen:
Man könnte auch eine Gefrierkurve fahren, indem man von Körpertemperatur bis auf 0°C herunterkühlt. Ein CD mit entsprechendem Peltier-Element ist aber schwer aufzutreiben.
An was und wie fest bindet das Spike-Protein?
Es wird aktuell spekuliert, dass das Spike-Protein möglicherweise neben dem ACE2-Rezeptor noch andere, sogenannte Co-Rezeptoren haben könnte.
Des Weiteren ist unklar wie fest das Impf-Spike-Protein an den ACE2 Rezeptor bindet und mit welchen Substanzen man es nachweislich wieder vom Rezeptor lösen kann.
Eine einfache und lange etablierte Messmethode stellt Biacore[13] bzw. Oberflächenplasmonenresonanzspektroskopie[14] dar. Damit ließe sich klären, ob und welches der Co-Rezeptor sein könnte (DPP4 steht im Verdacht) und ob das Spike den ACE2-Rezeptor zerstört oder wieder von diesem gelöst werden kann.
Messungen, die für Proteinfaltungsuntersuchungen wichtig wären
Was ansonsten zu bestimmen gewesen wäre, z. Bsp. über HX Pulse Labeling und NMR Spektroskopie[15], HX Pulse und MS Analyse und vieles Andere, womit ich keine Erfahrungen habe. Generell gilt da wohl, was Vendruscolo beschrieben hat:
Es ist daher von großer Bedeutung, die gut etablierte Charakterisierung der Struktur, Faltung und Stabilität nativer Zustände durch eine ähnliche Analyse der Struktur, des Aufbaus und der Stabilität anderer Zustände zu ergänzen - von ungefalteten und teilweise gefalteten Spezies, einschließlich nativ ungefalteter Zustände, bis hin zu aggregierten Spezies wie Amyloidfibrillen.[16]
Nichts von den oben erwähnten Experimenten wurde von BioNTech/Pfizer oder Moderna durchgeführt. Wir wissen nicht ansatzweise, womit wir es genetisch oder biochemisch beim Impf-Spike-Protein zu tun haben. Wir kennen seine grundlegenden Eigenschaften nicht ansatzweise.
ABER es wurden Milliarden von Menschen zu Produzenten eben dieses unbekannten Proteins mit zum großen Teil unbekannten Eigenschaften gemacht.
Labordaten, die von Wichtigkeit wären:
Die Nanolipide (ALC-0315 und ALC-0159) werden nicht nach Arzneimittelhandbuch hergestellt oder kontrolliert. Sie werden nach hauseigenen Regeln kontrolliert.
Welches sind diese Regeln, nach denen die Nanolipide geprüft werden?
Ich hätte gerne die HPLC Laufdateien (nicht die XLS-Daten, die Maschinendaten mit allen Sensoren, also z. Bsp. die Unicorn Dateien), um zu sehen, was sonst noch neben den Nanolipiden enthalten war und wie sauber diese Chemikalien tatsächlich waren, als sie im Menschen zu Einsatz kamen.
Massenspektrometrie des Schweißes von Geschlumpften wäre interessant. Sind Spikes oder Nanolipide im Schweiß nachweisbar?
Falls noch jemand Ideen hat, was an Analysen fehlt: Einfach in die Kommentare schreiben.
[1] Wikipedia-Autoren. (2021b). N1-Methylpseudouridin. de.wikipedia.org. https://de.wikipedia.org/wiki/N1-Methylpseudouridin
[2] https://www.tga.gov.au/sites/default/files/foi-2389-06.pdf
[3] McKernan, K., Helbert, Y., Kane, L. T., & McLaughlin, S. (2023, April 10). Sequencing of bivalent Moderna and Pfizer mRNA vaccines reveals nanogram to microgram quantities of expression vector dsDNA per dose. https://doi.org/10.31219/osf.io/b9t7m
[4] Ranzani M, Annunziato S, Adams DJ, Montini E. Cancer gene discovery: exploiting insertional mutagenesis. Mol Cancer Res. 2013 Oct;11(10):1141-58. doi: 10.1158/1541-7786.MCR-13-0244. Epub 2013 Aug 8. PMID: 23928056; PMCID: PMC3836224. https://pubmed.ncbi.nlm.nih.gov/23928056/
[5] Doerfler W. Adenoviral Vector DNA- and SARS-CoV-2 mRNA-Based Covid-19 Vaccines: Possible Integration into the Human Genome - Are Adenoviral Genes Expressed in Vector-based Vaccines? Virus Res. 2021 Sep;302:198466. doi: 10.1016/j.virusres.2021.198466. Epub 2021 Jun 1. PMID: 34087261; PMCID: PMC8168329. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8168329/
[6] DocCheck, M. B. (n.d.-d). Northern Blot - DocCheck Flexikon. DocCheck Flexikon. https://flexikon.doccheck.com/de/Northern_Blot
[7] DocCheck, M. B. (n.d.-f). Southern Blot - DocCheck Flexikon. DocCheck Flexikon. https://flexikon.doccheck.com/de/Southern-Blot
[8] ZyCoV-D becomes world’s first plasmid DNA vaccine for COVID-19. (2021, August 23). European Pharmaceutical Review. https://www.europeanpharmaceuticalreview.com/news/160968/zycov-d-becomes-worlds-first-plasmid-dna-vaccine-for-covid-19/
[9] DocCheck, M. B. (n.d.-b). FACS - DocCheck Flexikon. DocCheck Flexikon. https://flexikon.doccheck.com/de/FACS
[10] FreiDok plus - Nucleotide exchange and excision technology (NExT) DNA shuffling And Mutational studies on chloramphenicol actetyltransferase I (CATI). (n.d.-b). https://freidok.uni-freiburg.de/data/11610
[11] Wikipedia contributors. (2023b). Circular dichroism. Wikipedia. https://en.wikipedia.org/wiki/Circular_dichroism
[12] FreiDok plus - Nucleotide exchange and excision technology (NExT) DNA shuffling And Mutational studies on chloramphenicol actetyltransferase I (CATI). (n.d.-b). https://freidok.uni-freiburg.de/data/11610
[13] Cytiva. (n.d.). Biacore surface plasmon resonance - Cytiva. https://www.cytivalifesciences.com/en/us/support/online-tools/biacore-surface-plasmon-resonance
[14] Cytiva. (n.d.). Biacore surface plasmon resonance - Cytiva. https://www.cytivalifesciences.com/en/us/support/online-tools/biacore-surface-plasmon-resonance
[15] Englander SW, Mayne L. The nature of protein folding pathways. Proc Natl Acad Sci U S A. 2014 Nov 11;111(45):15873-80. doi: 10.1073/pnas.1411798111. Epub 2014 Oct 17. PMID: 25326421; PMCID: PMC4234557. https://pubmed.ncbi.nlm.nih.gov/25326421/
[16] Vendruscolo M, Knowles TP, Dobson CM. Protein solubility and protein homeostasis: a generic view of protein misfolding disorders. Cold Spring Harb Perspect Biol. 2011 Dec 1;3(12):a010454. doi: 10.1101/cshperspect.a010454. PMID: 21825020; PMCID: PMC3225949. https://pubmed.ncbi.nlm.nih.gov/21825020/





Ich habe eine Anmerkung und eine Frage. Die Anmerkung ist: Menschen sind zäh.
Ich bin ungeimpft aber 96% meiner geimpften Familie lebt noch, es geht ihnen jetzt nicht allen unbedingt ausgesprochen sehr gut, die Tante ist der blendet bei bei dem einen oder anderen ist das Immunsystem beeinträchtigt und ständige fieberhafte Erkrankungen sind die Folge, wir hatten einen plötzlich und unerwarteten Todesfall, Herz-Kreislauf versagen, Tod zusammengebrochen, 14 Tage nach der Impfung mit BionPech, keiner kann sagen was jetzt noch kommt in der Zukunft.
Nachdem ich meine Biologiekenntnisse im Bereich Zellaufbau und Funktion aufgefrischt habe und hier und bei anderen zur Gen Manipulation mitgelesen habe und dennoch nur mit einer sehr kleinen Taschenlampe ins Dunkle leuchte, frage ich mich wir halten das Wissenschaftler aus? Dieses Wissen und das Wissen, um die Konsequenzen.
Wie hält man diesen grenzdebienen Wahnsinn aus?
Ich werfe mal die Wörtchen "Post-translationale Modifikationen", "individuell-unterschiedliche Expression", "RNA-Regelungs-Kaskaden" (RNA kann mitunter selbst entscheiden, ob sie sich exprimieren lässt; bestimmende Faktoren gibts mannigfach; ggfs. wirkt sie aktivierend oder inhibierend auf irgendwelche Regelkreise, von deren Existenz man nach "aktuellem Kenntnisstand" (lol) noch nicht mal träumt), "Gen-Silencing" oder einfach nur "RNA-Ligase" ins Töpfchen. 2020 als der Hype einem überall vorne und hinten reingeschoben wurde, habe ich noch mit reverser Transkription (die Enzymmaschinerie dazu hat afaik jeder Zellkern) argumentiert.
Und wie schauts mit der Wirkung beliebig kurzer bzw. langer Peptid-Fragmente der Spikes aus? Sollbruchstellen drin?
Generell: Woher weiß ich, was alles in die RNA reinkodiert ist? Kann ja sein, dass da noch fröhlich andere Peptide exprimiert werden, die irgendwas machen: "Peptide-based inhibitor", "Müllerian inhibiting substance" (nur als Beispiel, was alles geht; ich sag nicht, dass das tatsächlich codiert ist).
Der Großteil der Mechanismen ist unbekannt. Und unbekannt, heißt eben unbekannt und nicht wünsch dir was.
Willkommen in der Empirie.
Du schreibst weiter vorne in der Reihe was von "Goldstandard Kristallstruktur". Das vergisst eine Klitzekleinigkeit: Das ist ne Festkörperstruktur. Proteine (and auch die meisten anderen Moleküle) "leben" aber in Lösung und haben da mitunter ganz andere Konformationen. Ohne Wasser faltet ein Protein teils völlig anders und es gibt nicht nur eine einzige Konformation, sondern mehrere, die ähnlich "energetisch" oder "thermodynamisch günstig" sind, je nachdem, wie die Umgebung ausschaut. Schmeiß das Protein doch mal in DMSO oder D2O und schau, wie es sich in diesem Solvens faltet. In ner Zelle hast du zwar Wasser als Lösemittel, aber in der Zelle geht es auch zu wie in der U-Bahn in Tokio. Will sagen: Da ist es gestopft voll: Andere Proteine, RNA, Wasser, organische und anorganische Ionen, Lipide, Aminosäuren und was da alles noch so rumsuppt bzw sich durchquetscht. Diese Umgebung kriegt niemand korrekt simuliert und berechnet. Erst recht nicht, weil das alles ja auch noch dynamisch ist. Deswegen sind Kristallstrukturen schön, um eine Verbindung zu charakterisieren und die Konnektivität (welches Atom hängt an welchem anderen Atom) nachzuweisen. Aber für Konformationen in Lösung, in Dynamik beweisen die GAR NIX. Ist wie ein Foto von einem sitzenden Pferd. Davon dann abzuleiten, dass es nicht rennen kann, wäre blödsinnig.