Ugurs grenzdebile Schwachsinnsideen im Protein Design
Teil 4c: Schon mal was vom Epitranskiptom gehört, Ugur?
Da es sich bei diesem Artikel um die zweite Fortsetzung zu Teil 4 der Serie handelt, wird das Grundwissen der vorherigen Teile vorausgesetzt, auch wenn sich Einiges wiederholt.
Am 21. Januar 2020 erschien ein interessantes Paper mit dem Titel „Eine Perspektive auf molekularer Ebene zur Häufigkeit, Verteilung und den Folgen von Veränderungen der mRNA“
Jones JD, Monroe J, Koutmou KS. A molecular-level perspective on the frequency, distribution, and consequences of messenger RNA modifications. Wiley Interdiscip Rev RNA. 2020 Jul;11(4):e1586. doi: 10.1002/wrna.1586. Epub 2020 Jan 21. PMID: 31960607; PMCID: PMC8243748. https://pubmed.ncbi.nlm.nih.gov/31960607/
Dieses Paper hätten die modRNA-Gläubigen lesen können, bevor sie dieses System auf die Menschheit losgelassen haben, da hätten sie nämlich erkennen können, dass sich dar gerade eine vollkommen neue Richtung der Biologie entwickelt namens Epitranskiptomik, die gerade gezeugt wurde und nicht mal so wirklich in den Kinderschuhen steckt. Vielleicht wäre ihnen dann aufgefallen, dass diese modRNA-Plattform so gar keine gute Idee ist.
Ich werde in diesem Artikel das Paper in gekürzter, kommentierter Form auf die Highlights zusammenfassen, die dem interessierten Laien erkenntlich machen sollten, dass die modRNA-Plattform auch aus Epitraskriptomik-Sicht eine grenzdebile Schwachsinnsidee ist und das hätte man schon im Januar 2020 erkennen können/sollen.
Da ist ein Review, Herrgott, seichte wissenschaftliche Unterhaltungsliteratur, die man zur Entspannung liest. Das sollte man auch in einer Pandemie auf dem Klo hinbekommen. Das hat bei der Sumpfsuppe doch auch so super funktioniert.
Ich werde hierfür das Paper hauptsächlich übersetzen, kürzen und mit ein paar ironischen Kommentaren versehen, um die wichtigsten Stellen so zu überspitzen, dass selbst das dümmste Schaf in der Überspitzung zumindest die Grundlagen mitnehmen sollte.
Abstract
„Jüngste Fortschritte bei den genomweiten Sequenzierungstechnologien haben die Identifizierung von mRNA-Modifikationen als potenzielles neues Grenzgebiet der Genregulierung ermöglicht und zur Entwicklung des Epitranskriptom-Bereichs geführt. In der Folge wurden zahlreiche bahnbrechende Entdeckungen gemacht, darunter neue Modifikationen, nukleosemodifizierende Enzyme ("writers" und "erasers") und RNA-bindende Proteine, die chemische Modifikationen erkennen ("readers").“
Wir haben hier also schon im Abstract ein paar gute aber auch ein paar schlechte Nachrichten.
Die gute Nachricht zuerst: Es gibt die Hypothese, dass es Proteine gibt, die diese modRNA Modifikationen rückgängig machen können. Diese Proteine heißen „eraser“. Leider, leider handelt es sich dabei um eine Hypothese, denn bisher konnte noch keines dieser Eraser-Proteine für das in der Sumpfsuppe verwendete modifizierte Nucleodid (m1Ψ) identifiziert werden, aber immerhin für m6A, m1A un m5C: „Bis heute wurden keine "reader" oder "eraser" für andere mRNA-Modifikationen (als m6A, m1A und m5C) gemeldet, obwohl vorgeschlagen wurde, dass das Ribosom als universelles Lesegerät für Modifikationen in mRNA-kodierenden Regionen dienen kann“. ABER es muss sie wohl definitiv geben. Sooooooo weit ist man im Fachbereich der Epitranskriptomik, man hat zumindest schon einmal Hypothesen, die man nun bearbeiten kann.
Die schlechte Nachricht: „Diese Entdeckungen öffneten die Tür zum Verständnis, wie posttranskriptionale mRNA-Modifikationen den mRNA-Lebenszyklus modulieren können, und stellten eine Verbindung zwischen dem Epitranskriptom und menschlicher Gesundheit und Krankheit her.“
Die Tür zum Verständnis wurde geöffnet. Man hat also erkannt, oh FCK, das Epitranskiptom kann Ursache für Krankheit sein.
Was Du nicht sagst Sherlock.
Es wird aber noch besser und wir sind erst im Abstract:
„Obwohl ihre Bedeutung immer mehr erkannt wird, sind grundlegende Fragen zur Identität, Verbreitung und zu den funktionellen Folgen von mRNA-Modifikationen noch nicht geklärt.“
Und weil das alles noch komplett unbekannt und NICHT GEKLÄRT ist, haben Moderna und BioNTech/Pfizer einfach mal alles Uracile (U) gegen N1-Methylpseudouridin (m1Ψ) ausgetauscht, so um auf der sicheren Seite zu sein, schätze ich.
„Im Zuge des Fortschritts auf diesem Gebiet sehen wir die Notwendigkeit einer weiteren Integration von quantitativen und reduktionistischen Ansätzen zur Ergänzung von transkriptomweiten Studien, um einen Rahmen auf molekularer Ebene für das Verständnis der Auswirkungen chemischer Veränderungen der mRNA auf biologische Prozesse zu schaffen.“
Also, verstehe ich das richtig, man ist erst dabei dieses Fachgebiet überhaupt zu etablieren und Studienansätze zu entwickeln, damit man die Epitraskriptomik erforschen kann?!
Wie gut, dass man das nun in Milliarden von Menschen live beobachten kann, welchen Einfluss diese Modifikationen in mehrzelligen Euraryoten auf Gesundheit und Krankheit haben, da kann man direkt mit einem ordentlich großen Datensatz starten und kann auf die Tierversuche verzichten.
Der geneigte Leser, der zwischen den Zeilen lesen kann, erkennt sicherlich die Stimmung, in welcher ich mich beim Schreiben befinde. Gummizelle, damit ich den Kopf gegen die Wand rammen kann, ohne Schaden zu nehmen, wäre aktuell echt wünschenswert, Rauputz tut so weh.
Einleitung
„[…] mRNA-Modifikationen haben das Potenzial, die Proteinexpression zu steuern […]
Es ist noch unklar, ob die meisten Modifikationen auf Off-Target-Aktivitäten auf ncRNA-modifizierende Enzyme zurückzuführen sind, oder ob sie eine neue Ebene der posttranskriptionellen Kontrolle darstellen. Unabhängig davon haben mRNA-Modifikationen wahrscheinlich biologische Konsequenzen, da diese chemischen Markierungen die Interaktionen zwischen mRNAs und der zellulären Maschinerie beeinflussen können. Die Untersuchung von mRNA-Modifikationen ist ein schnell wachsendes Gebiet, da die Forscher versuchen, den Einfluss von mRNA-Modifikationen auf die Biologie und die menschliche Gesundheit zu ermitteln. […]
Um zu verstehen, wie der Status der mRNA-Modifikation zu einzelnen biologischen Prozessen und Krankheitszuständen beiträgt, müssen jedoch noch wichtige grundlegende Fragen zum Einbau und zu den Folgen der mRNA-Modifikation auf molekularer Ebene untersucht werden.“
Erst einmal ein wenig Begriffserklärung:
ncRNA = non coding RNA, also RNA, die nicht für Proteine kodiert, sondern teils auch regulatorische und andere Funktionen hat. Die Anzahl der ncRNA im Menschen ist unbekannt. Und von vielen der identifizierten ncRNAs kennt man ihre Funktion nicht. Einige kennt man aber wie tRNA (transfer RNA) oder rRNA (ribosomale RNA), daneben gibt es aber noch ein paar mehr, von denen ich bisher teilweise auch noch nie gehört habe: microRNAs, siRNAs, piRNAs, snoRNAs, snRNAs, exRNAs, scaRNAs und lange ncRNAs (Xist und HOTAIR (lustiger Name übrigens hot air = heiße Luft)). Muss man nicht im Detail verstehen, soll nur zeigen, da ist noch viel unbekannt, man weiß aber, da gibt es was und das scheint irgendwie wichtig zu sein, will man gesund bleiben.
Off-Target-Aktivitäten = Effekte wo anders als sie eigentlich stattfinden sollten. Also wenn z. Bsp. die Sumpfsuppe nicht im Muskel verbleibt und im ganzen Körper diversen Schabernack treibt.
Im Großen und Ganzen sagt die Einleitung, wir haben keine Ahnung von nichts. Nicht einmal ob es da noch Proteine gibt, die Einfluss auf die modRNA nehmen. Wie auch immer, das hat alles wahrscheinlich biologische Konsequenzen (wer hätte das gedacht?). Es sind nicht mal die grundlegenden Fragen zum Einbau der Modifikationen geklärt, ABER Ugur hat einfach mal alle U gegen m1Ψ ausgetauscht. Wer braucht schon die Klärung von grundlegenden Fragen oder auch nur ein grobes Verständnis über die Funktion dieser Modifikationen?! Ugur bestimmt nicht, denn Ugur ist ein Genie und weiß es natürlich besser.
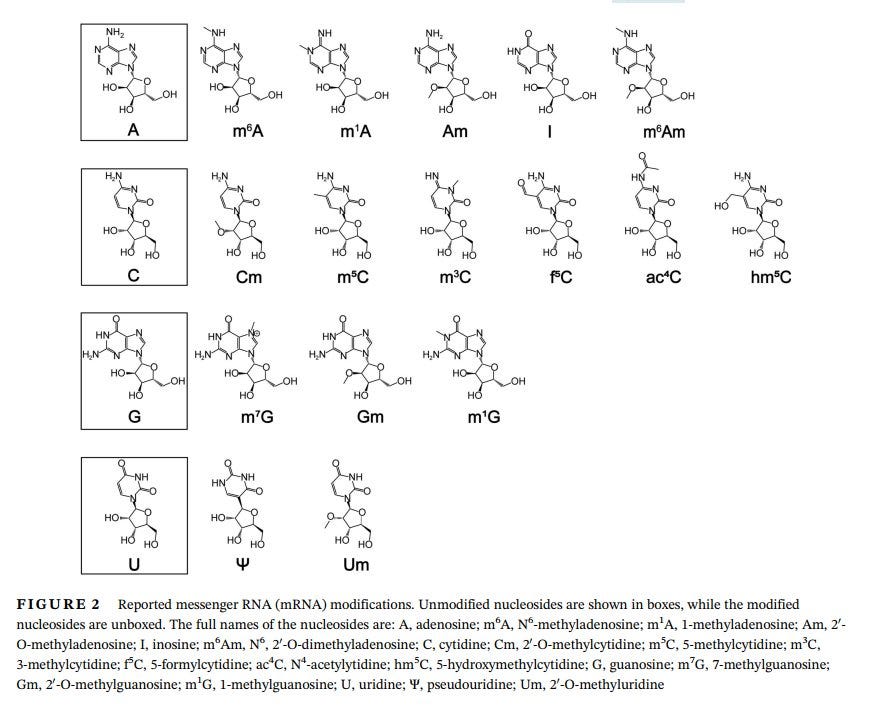
Bei der nun folgenden Abbildung 2 im Paper fällt einem etwas unangenehm auf:
Da fehlt doch was?
Na, was fehlt?
m1Ψ fehlt.
Überhaupt glänzt m1Ψ in diesem Paper Großteils durch Abwesenheit, weil es dazu wohl so wenig Daten gibt, dass es nur in einem Nebensatz erwähnt wird.
Welches Nucleotid hat man genommen, daher sicherheitshalber genommen? Eines bei dem viele Daten vorliegen oder eines, das so wenig Daten hat, dass man es in einem Review zum Thema nur am Rande erwähnt?!
Einmal mit Profis arbeiten, sage ich da nur.
Gut, die Daten werden jetzt nun wohl im großangelegten Menschenversuch um Nachhinein erzeugt und werden noch viele Reviews füllen, schätze ich.
Ab dieser Stelle konzentriert sich der Autor hauptsächlich auf ein modifiziertes Nucleotid namens m6A, weil
„wir haben wir ein begrenztes (oder gar kein) Bild von der biologischen Rolle der anderen 14 gemeldeten mRNA-Modifikationen.“
Und wie erwähnt, m1Ψ zählt da nicht dazu, das ist nicht mal auf der Übersicht, so „viele“ Daten hat man dazu.
QUANTITATIVE ANSÄTZE ZUR UNTERSUCHUNG DER MRNA-VERÄNDERUNG: EBENEN UND FOLGEN
In diesem Teil der Publikation widmet sich der Autor den eigentlich noch nicht wirklich etablierten Nachweismethoden dieser RNA-Modifikationen.
Man kann Massenspektrometrie machen, nachdem man die modRNA zerlegt hat, und so quantifizieren, was und wieviel drinnen ist, ABER
„MS-Ansätze sind begrenzt, da sie keinen Sequenzkontext für modifizierte Nukleoside liefern und große Mengen hochgereinigter mRNA erfordern.“
Im Klartext: Man weiß, dass es drinnen ist, aber nicht wo und warum und das auch nur für m6A und nicht für m1Ψ. Da hat man nicht mal diese Methodik etabliert.
Ähm…. Wie genau haben Moderna und BioNTech dann bitte ihr Endprodukt charakterisiert, wenn man das noch gar nicht kann?
Ich frag nur für einen Freund.
„In ähnlicher Weise können die Halbwertszeiten aller zellulären mRNAs parallel gemessen werden, indem RNA-seq verwendet wird, um die zeitabhängige Verringerung der mRNA-Spiegel nach Abschaltung der Transkription durch kleine Moleküle wie Actinomycin oder temperatursensitive RNA-Polymerase-II-Mutanten zu beobachten Ribosomen-Profiling und transkriptomweite Halbwertszeitmessungen liefern aussagekräftige Informationen über die Details der mRNA-Stabilität und -Translation und haben unser Verständnis der RNA-Biologie erheblich verbessert.“
Wie genau haben Moderna und BioNTech/Pfizer die Halbwertszeit ihrer modRNA bestimmt und wie verlässlich sind diese Daten? Wenn man zugrunde legt, was in den Behandelten Menschen an Messwerten aktuell erhoben wird, scheinen mir die so erhobenen Daten zur Halbwertszeit der modRNA nicht sonderlich verlässlich zu sein.
In-vitro-Biochemie und Strukturbiologie
„Generell ist über die Strukturen von RNAs im Vergleich zu Proteinen viel weniger bekannt. Dies wird durch die Tatsache belegt, dass Strukturen von RNA und RNA-Protein-Komplexen <5 % der in der Protein Data Bank (PDB) hinterlegten Strukturen ausmachen.“
Die schönen Bildchen, welche Strukturen von RNA miteinander interagieren und diese netten Muster bilden sind MODELLE!!! Nur so nebenbei. Keiner weiß, wie RNA wirklich aussieht, zumal diese MODELLE 2D sind und RNAs ein 3D Struktur haben.
Bei Proteinen würde das bedeuten, man weiß welche Aminosäuren verbaut sind und kann vorhersagen wo Helixes oder Faltblatt Strukturen sein müssten, wie diese sich im Raum anordnen weiß man nicht.
Man hat keinen Schimmer, wie sich die RNA Strukturen im 3D Raum anordnen. Noch weniger Ahnung, als bei Proteinen, weil es noch weniger Strukturen gibt. Aber selbst in der 2D Ebene fällt auf, dass da was gehörig schiefgelaufen ist. Da braucht man von 3D gar nicht erst anfangen[i].
Und da hat man reingepfuscht, ohne Sinn und Verstand.
„Der Vergleich von modifizierten und nicht modifizierten mRNAs kann zusätzliche Informationen darüber liefern, wie die Form von mRNAs durch Modifikationen beeinflusst wird.“
Verstehe ich das richtig? Das hat man bisher noch nicht gemacht?! Nicht einmal bei Moderna oder BioNTech/Pfizer? Wäre das nicht sinnvoll, um zu sehen, inwieweit sich modRNA vom viralen Original unterscheidet. DAS wäre ja noch OHNE „Codonoptimierung“, die käme noch hinzu, so ganz nebenbei.
„Strukturstudien werden sich nicht nur als nützlich erweisen, um zu verstehen, wie sich Modifikationen auf die intrinsischen Eigenschaften von mRNAs auswirken, um deren Funktion und Stabilität zu beeinflussen, […]“
Im Original steht da „will prove“, also eindeutig ein Futur. Also irgendwann in der Zukunft, werden wir vielleicht Strukturstudien haben, die zeigen, wie die jetzt schon in der Plörre verwendeten Modifikationen die Struktur, Funktion, Stabilität und ganz generell die Eigenschaften der modRNA im weitesten Sinne beeinflusst haben.
Das schafft doch Vertrauen in die gerade gestartete Impfkampagne mit den angepassten Plörren.
„Es ist davon auszugehen, dass Veränderungen in der mRNA-Struktur, der Ladung oder der Fähigkeit zur Basenpaarung die Interaktion dieser Moleküle mit anderen Biomolekülen verändern. Daher sind kinetische und thermodynamische In-vitro-Tests erforderlich, um festzustellen, wie Modifikationen die Wechselwirkungen zwischen mRNA und Proteinen, anderen RNAs oder dem Ribosom verändern.“
Also diese Daten habe ich weder von Moderna noch von BioNTech/Pfizer in den bisher veröffentlichten Datensätzen finden können. Vielleicht kommt da ja noch irgendwann was nach?
Neeee, wohl eher nicht.
Es war also im Januar 2020 bereits davon auszugehen, dass die Änderung der mRNA-Struktur zu einer modRNA-Struktur die Interaktion mit anderen Biomolekülen im weitesten Sinne verändern wird (und ich setze hier mit Absicht kein komme, um den Satz mehrdeutig zu lassen).
Könnte es sein, dass Probleme verursacht wurden, von denen wir nicht einmal wissen, dass sie existieren, weil wir nicht wissen, dass es da überhaupt Probleme geben kann?
Hat man das in den klinischen Studien bedacht?
Vermutlich nicht?
“Solche Studien werden von entscheidender Bedeutung sein, wenn die Forscher beginnen, die Art und das Ausmaß der Auswirkungen von mRNA-Veränderungen auf die Biologie zu verstehen.“
Ja, ganz besonders, wenn sie selbst mehrfach mit dem Zeugs geschlumpft sind, da bekommt der Begriff, der im Original verwendet wird, eine ganz andere Bedeutung, denn da steht auf Englisch: „such studies will be vital“. Vital = lebenswichtig und nicht nur von entscheidender Bedeutung.
Wenn das mal nicht ein Ansporn ist ein paar Wochenenden im Labor einzulegen und ein bisschen länger zu arbeiten, dann weiß ich auch nicht.
AKTUELLE QUANTITATIVE PERSPEKTIVE AUF mRNA-ÄNDERUNGEN
„Zusammengenommen deuten diese Daten darauf hin, dass mehrere mRNA-Modifikationen, die relativ häufig vorkommen, die mRNA-Struktur beeinflussen und die Art und Weise beeinflussen können, wie Proteine und das Ribosom mit Transkripten interagieren.“
Ich glaube, dieser Satz ist selbsterklärend und sehr einfach zu verstehen, selbst für Ugur. Der hat ihn aber leider nicht gelesen, schätze ich.
In diesem Kapitel geht es dann wieder hauptsächlich um m6A, weil es für die anderen modifizierten Nucleotide kaum Daten gibt.
Aber, die Modifikationen scheinen konserviert zu sein und man weiß wenig darüber wie oft sie in bestimmten mRNAs verbaut werden. Von Ψ, mit dem Karikó gearbeitet hat, weiß man, dass es sehr häufig verbaut wird: „Zusammengenommen deuten diese Daten darauf hin, dass die Häufigkeit des Einbaus von Ψ, zumindest an einigen Stellen, wahrscheinlich hoch ist.“
Nur Dummerweise hat man m1Ψ verbaut, und davon weiß man das halt nicht.
Hätten sie nur Ψ genommen, da hätte man wenigstens ein bisschen mehr Daten als für m1Ψ.
Aber das wäre nicht so intellektuell „aufregend für die Forscherseele“ (frei zitiert nach Project Lightspeed S. 101) unseres Ugur gewesen.
Auswirkungen von mRNA-Veränderungen auf die Übersetzung
„Die Entschlüsselung der Auswirkungen von mRNA-Veränderungen auf die Translation in biologischen Systemen war aus mehreren Gründen eine Herausforderung. […] Die Heterogenität bei der Belegung von Modifikationen erschwert es den Forschern, die Translation modifizierter Transkripte in vivo direkt zu beobachten. Schließlich kann es problematisch sein, die Auswirkungen der Translation von der Protein- und mRNA-Stabilität auf die Proteinproduktion in Zellen vollständig zu entschlüsseln. So sind reporterbasierte Studien zu widersprüchlichen Schlussfolgerungen darüber gelangt, wie verschiedene Modifikationen die Translation beeinflussen. […]
Erste In-vitro-Studien mit unterschiedlicher Auflösung zu einer begrenzten Anzahl von mRNA-Modifikationen deuten darauf hin, dass die Modifikationen die Gesamtgeschwindigkeit und Zuverlässigkeit der Proteinsynthese beeinflussen […]
Der Schweregrad des Defekts bei der Proteinexpression hängt in hohem Maße von der Position der Veränderung innerhalb eines Codons ab - die Proteinausbeute kann je nach Position einer Veränderung innerhalb eines einzelnen Codons um das 25-fache schwanken. […]
Diese Veränderungen wirken sich auf mehrere Schritte des kinetischen Weges der Ribosomen aus. […]
Weitere Arbeiten müssen durchgeführt werden, um die Beziehung zwischen den unterschiedlichen Beobachtungen bei In-vitro- und Ribosomen-Profilierungsstudien in Einklang zu bringen.
Da Modifikationen die grundlegenden Eigenschaften von RNAs, einschließlich ihrer Sekundärstrukturen und Basenpaarungsfähigkeiten, verändern können, wurde vorgeschlagen, dass eine Folge der mRNA-Modifikation darin bestehen könnte, den Einbau mehrerer Aminosäuren an einem einzigen Codon zu fördern. […]
mRNA-Veränderungen können sowohl die Geschwindigkeit als auch die Treue der Translation beeinflussen. Wie diese Veränderungen zur Biologie beitragen, ist noch nicht geklärt.“
Im Klartext, sehr viele Worte, um zu beschreiben, was man alles nicht weiß, statt zu sagen, dass man eigentlich noch gar nichts weiß.
“Eine Folge der mRNA-Modifikation darin bestehen könnte, den Einbau mehrerer Aminosäuren an einem einzigen Codon zu fördern.”
DAS wurde letztendlich in einem Nature Paper auch für die Plörre belegt [ii], bzw. dass es durch die Modifikationen zumindest zu Rasterschubmutationen kommt, wenn auch nicht direkt zum Einbau mehrerer Aminosäuren an einem Codon. D.h. durch einen Lesefehler wegen der mRNA-Modifikationen springt das Ribosom von Leseraster 1 in Leseraster 2, in welchem sich bei der Plörre leider kein Stopp-Codon befindet, wie man das eigentlich gelernt hätte, hätte man Ahnung von Protein Engineering.
8% der Proteine waren betroffen und 1/3 der Geschlumpften hatten entsprechende alternative Protein anhand einer Antikörperanalyse. Bei Astra Geschlumpften passierte das NICHT, es liegt also an der modRNA.
Und genau davor hat die Nobelpreisträgerin Karikó übrigens auch gewarnt, man kann es kaum glauben:
EN: Potent cryptic T cell epitopes may be generated when the IVT mRNA is translated in different frames owing to ribosomal frame-shifting or when translation is initiated either internally or from a CUG start codon. [iii]
DE: Potente kryptische T-Zell-Epitope können entstehen, wenn die IVT-mRNA aufgrund von ribosomalem Frame-Shifting in verschiedenen Frames translatiert wird oder wenn die Translation entweder intern oder von einem CUG-Startcodon aus eingeleitet wird.
Man kann jetzt nicht wirklich behaupten, dass das komplett überraschend käme, zumal aus den EMA Unterlagen bereits klar war, dass etwas mit der Größe des Spike-Proteins nicht stimmen konnte.
Ugurs grenzdebile Schwachsinnsideen im Protein Design
Eine der bekanntesten und klassischsten Formen der Propaganda oder Meinungsmanipulation ist die Methode der Auslassung oder verkürzten Erzählung. Man lügt nicht direkt, man lässt jedoch für die Meinungsbildung wichtige Informationen weg. Diese Methode der Manipulation wurde bei der Geschichte der Verwendung von N1-Methylpseudouridin angewandt.
Zusätzlicher Verbau von Aminosäuren in Proteine dürfte ebenfalls sehr „interessante“ Effekte auf die Struktur haben, schätze ich? Wurde bisher aber noch nicht nachgewiesen.
Sogar die verschiedenen Messungen widersprechen sich in ihren Ergebnissen. Es gibt nicht einmal etablierte Messmethoden, die zu einem eindeutigen Ergebnis führen würden.
Wie genau wurden entsprechende Studien in Zellkultur für die Plörre dann von Moderna und BioNTech/Pfizer durchgeführt? Möglicherweise gar nicht?!
DAS haben dann Wissenschaftler aus der University of Cambridge in UK für BioNTech/Pfizer erledigt.
Änderungen der mRNA-Protein-Wechselwirkungen
„Viele RNA-bindende Proteine interagieren mit einer Vielzahl von mRNA-Sequenzen, und selbst kleine Störungen der Affinität haben das Potenzial, das zelluläre Umfeld und damit das Schicksal einer Vielzahl von mRNAs zu verändern“
Wie genau beeinflusst dabei die „Codonoptimierung“ und das Ersetzen von U mit m1Ψ diese RNA-bindenden Proteine? Könnte es da zu Störungen durch Fehlerkennungen kommen?
„Trotz der Entdeckung mehrerer Proteine, die mit modifizierten mRNAs interagieren, ist weniger klar, inwieweit Modifikationen die mRNA-Protein-Interaktionen verändern.“
Na dann ist ja gut.
Conclusion
„Der Bereich der Epitranskriptome schlägt schnell ein neues Kapitel auf, indem er durch die Entdeckung von Modifikationen die biologischen Rollen und Mechanismen einer breiten Palette von mRNA-Modifikationen untersucht. […]
Der nächste Horizont für das aufstrebende Feld der mRNA-Modifikation ist die Etablierung einer Sichtweise auf molekularer Ebene, wie Modifikationen die Interaktionen zwischen mRNAs und der zellulären Maschinerie verändern. […]
Die Möglichkeit, die Interaktionen von vollständig modifizierten/unmodifiziertenmRNAs mit gereinigten Komponenten zu vergleichen, wird besonders wertvoll sein, wenn man bedenkt, wie schwierig es ist, transkriptomweite Kartierungs-, Halbwertszeit- und Ribosomenprofilierungsdaten für heterogene Populationen von substöchiometrisch modifizierten mRNAs zu interpretieren. […]
Transkriptomweite Studien werden letztlich erforderlich sein, um die biologischen Folgen des Epitranskriptoms zu ermitteln.“
Eigentlich hätte Ugur nur die Conclusion lesen müssen, um zu erkennen, ganz blöde Idee, was er da abziehen will.
DAS waren nur meine „Highlights“ aus der Publikation. Wer es genau wissen will, muss halt das Original komplett lesen.
Der WHO ist das Problem mit den unnatürlichen dNTPs bekannt:
*reg-considerations-on-rna-vaccines_1st-draft_pc_tz_22122020.pdf
[i] McKernan, K., Kyriakopoulos, A. M., & McCullough, P. A. (2021, November 25). Differences in Vaccine and SARS-CoV-2 Replication Derived mRNA: Implications for Cell Biology and Future Disease. https://doi.org/10.31219/osf.io/bcsa6
[ii] Mulroney TE, Pöyry T, Yam-Puc JC, Rust M, Harvey RF, Kalmar L, Horner E, Booth L, Ferreira AP, Stoneley M, Sawarkar R, Mentzer AJ, Lilley KS, Smales CM, von der Haar T, Turtle L, Dunachie S, Klenerman P, Thaventhiran JED, Willis AE. N1-methylpseudouridylation of mRNA causes +1 ribosomal frameshifting. Nature. 2023 Dec 6. doi: 10.1038/s41586-023-06800-3. Epub ahead of print. PMID: 38057663. https://pubmed.ncbi.nlm.nih.gov/38057663/
[iii] Sahin U, Karikó K, Türeci Ö. mRNA-based therapeutics--developing a new class of drugs. Nat Rev Drug Discov. 2014 Oct;13(10):759-80. doi: 10.1038/nrd4278. Epub 2014 Sep 19. PMID: 25233993. https://pubmed.ncbi.nlm.nih.gov/25233993/







Hallo Frau Dr. Stebel! Ich werde diesen Beitrag bei DoorlessCarp verlinken, aber vielleicht versuchen Sie auch selbst mit DoorlessCarp in Kontakt zutreten. Auf dessen Substack finden sich immer sehr ausführlich recherchierte Reviews von Wissenschaftlichen Arbeiten.
Beim ersten überfliegen habe ich ein paar Rechtschreibfehler und so gefunden.... aber da ich selbst sehr oft Fehler in meinen Beiträgen habe, weiss ich nicht ob Ich sie eigentlich darauf hinweisen darf =8)
Ansonsten, bitte weiter so! Ihre Sichtweise auf die katastrophalen Versäumnisse bei der Entwicklung dieser "Sumpfsuppen" ist meiner Ansicht nach von sehr großer Bedeutung. Und je mehr Hin- und Beweise in der Öffentlichkeit zusammengetragen werden, das die "Impfkampagnen" unvorhersehbare Folgen hatten und haben ist von enormer Bedeutung.
Ich krieg schon den Würgreiz, wenn ich daran denke, das verschiedene Personen schon davon fabuliert haben diese modRNA/LNP Technologie für einen Nobelpreis vorzuschlagen... leider hat Ugur ja schon etliche andere Preise übergeben bekommen. Meiner Ansicht nach, sollten diese vielleicht in der 3mx3m Zelle zusammen mit Ugur bis ans Ende seiner natürlichen Lebensspanne weggesperrt werden.
Liebe Sabine,
bitte mal das hier zu Gemüte führen...
Impfstoffe und Krebszelllinien: https://hoch2.tv/sendung/230913-horizont/
Viele Grüße
Fabian