Ugurs grenzdebile Schwachsinnsideen im Protein Design
Teil 1 Warum es eine grenzdebil schwachsinnige Idee war, die Codons zu „optimieren“ #COptiGate
Wenn man als Doktorand im Labor anfängt, muss man sich in sein Projekt erst einmal einlesen. Gewöhnlich nutzt man dazu zunächst einmal sogenannte Übersichtsartikel (Reviews), die einem, einen Überblick über das bereits vorhandene Wissen verschaffen. Man muss das Rad schließlich nicht neu erfinden, sondern Neues erforschen, und dazu muss man verstanden haben, was an Wissen bereits vorhanden ist.
Übersichtsartikel werden auch als „Grauliteratur“ bezeichnet. Sie sind in gewisser Weise die „Unterhaltungsliteratur“ in der Wissenschaft, mit welcher man sich gemütlich und meist in einfacherer Sprache, einen soliden Überblich über ein Fachgebiet anlesen kann, ohne allzu viel Recherchearbeit investieren zu müssen. Reviews sind so eine Art Lektürehilfe/Lektüreschlüssel für Wissenschaft. Genau wie Schüler gerne nur die Lektürehilfe zu Faust lesen, statt das Theaterstück, lesen Wissenschaftler lieber Reviews statt vieler, vieler einzelner Paper, aus denen man sich dann die kleinen Puzzlestücke auch noch selbst zusammenstückeln muss und wie beim Theaterstück häufig auch noch zwischen den Zeilen lesen muss. Das ist beim Review von jemandem anderen für einen erledigt worden. 60 Jahre Forschung auf 10 Seiten, so macht das Spaß. Will man etwas genauer wissen, kann man in die Endnoten gehen und sich die Details genauer anschauen.
Arbeitet man auf einem eng beackerten Wissenschaftsfeld, gibt es viele schöne Reviews, aber auch viel Konkurrenz. Arbeitet man auf einem verwaisten Gebiet, kann es sein, dass man selbst der Depp ist, der das Review schreiben muss, weil man sich ja gerade so schön in die Literatur eingearbeitet hat und der Laborleiter (auch PI, primary investigator genannt) das eine gute Idee findet, wenn das mal einer wieder hübsch zusammenfasst. Das erwischt zugegebenermaßen normalerweise nicht den frischen Doktorenden, sondern den frischen, neuen Post-Doc (also jemand, der nach der Doktorarbeit in einer neuen Gruppe seine wissenschaftliche Arbeit 6 Jahre fortsetzt, um anschließend eine Habilitationsstelle zu suchen und den steinigen Weg zur Professur anzutreten) bzw. den Doktoranden in den letzten Zügen seiner Promotion. Wenn man ohnehin dabei ist zusammenzuschreiben, kann man auch direkt ein Review verfassen, man ist ja ohnehin gerade dabei.
Ich verlange von Ugur nichts anderes als das, was ich von einem frischen Doktoranden erwarten würde, wenn er sich in ein neues Themengebiet einarbeitet. Denn es war ja PANEMDIE!!! Wir werden alle sterben!!! Und Ugur war im Stress. Vielleicht hat er ja wirklich daran geglaubt, wer weiß. Für ein paar Reviews zum Thema Proteinoptimierung und Proteinfaltung hätte die Zeit aber definitiv gereicht. Dann hätte Ugur, sein Team oder vielleicht jemand bei Pfizer ein paar fatale und extrem peinliche Anfängerfehler vermeiden können.
Hier nun beginne ich mit einer kleinen Reihe über die Anfängerfehler im Proteindesign, die Ugur unterlaufen sind.
BioNTech/Pfizer und Moderna haben sich verhalten wie ein übermütiger, junger, betrunkener Autofahrer, der keinen Führerschein hat und daher die Straßenregeln nicht kennt (=Regeln des Proteindesigns), sein Auto nicht unter Kontrolle hat und mit 200kmh/h durch eine volle Fußgängerzone zur Haupteinkaufzeit am Samstag rast.
Einen solchen Autofahrer von seiner Schuld freizusprechen, weil er ja keinen Führerschein habe und die Regeln somit nicht kennen konnte, würde wohl kaum einem Beteiligten in den Sinn kommen.
Im Folgenden werde ich die „Verkehrsregeln“ beschreiben und erklären, die BioNTech/Pfizer und Moderna möglicherweise nicht kannten oder fahrlässig ignoriert haben, um sich anschließend in diversen Veröffentlichungen auch noch damit zu brüsten, dass sie eben genau diese Fehler begangen haben.
Ich werde jedem seiner Fehler einen eigenen Substack widmen, damit man sie jeweils einzeln in ihrer vollen Schönheit betrachten und würdigen kann. Teilweise passierten die Fehler schon auf einem sehr grundsätzlichen Niveau des Schülerdudens Biologie. Da ich aber davon ausgehe, dass Ugur keinen aktuelleren Schülerduden besitzt, werde ich mit Reviews von vor Corona argumentieren, die er hätte lesen können und auch lesen müssen, bevor er Hand an etwas gelegt hat, das weder er noch die Forscher, die auf diesem Gebiet seit Jahrzehnten arbeiten, verstanden haben. Wenn es die Experten nicht verstehen, dann lässt man als Laie einfach mal die Finger davon, wäre mein unqualifizierter Vorschlag in diesem Zusammenhang.
Wäre Ugur einer meiner Studenten gewesen, ich hätte ihn durch die Prüfung fallen lassen. Und dabei bewegen wir und hier „nur“ auf dem Gebiet der Codonoptimierung. Vom N1-Methylpseudouridin habe ich hier noch gar nicht angefangen, DAS ist auch so eine grenzdebile Schwachsinnsidee, der ich einen anderen Substack widmen werde. Ganz abgesehen davon, dass man die Finger von einem Protein lässt, dass ohnehin schon in einem labilen strukturellen Gleichgewicht ist und dann vor allem in so ein Protein keine Proline verbaut.
Aktuell sieht die Planung für diese Substack-Reihe wie folgt aus:
Teil 1: Warum es eine grenzdebil schwachsinnige Idee war, die Codons zu „optimieren“.
Teil 2: Warum es eine grenzdebil schwachsinnige Idee war, Proline zu verbauen.
Teil 3 Warum es eine grenzdebil schwachsinnige Idee war, ein strukturell dynamisches Protein überhaupt auch nur anzufassen, dessen Struktur man nicht einmal im Detail kennt.
Teil 4: Warum es eine grenzdebil schwachsinnige Idee war, N1-Methylpseudouridin zu verwenden.
Teil 5: Welche biochemischen Basisdaten des Spikes ich gerne hätte also meine persönliche Analysewunschliste
Wenn das jetzt einen irgendwie genervt, übellaunigen, angefressenen Eindruck macht, dann ist der Eindruck vollkommen korrekt. Ich habe nach der Recherche zu dieser debilen Schwachsinnsidee seitens Ugurs bereits 2 Tage schlechte Laune und muss die nun los werden.
Beginnen wir einmal damit, was Ugur selbst zum Optimierungsproblem in seinem Buch „Project Lightspeed“ geschrieben hat und was ich in meiner Reihe „Einmal mit Profis arbeiten“ bereits dazu angedeutet habe, ohne ins Detail zu gehen, das mache ich nämlich in dieser Reihe.
„Die Nukleotidsequenz für den knubbeligen Stachel war leicht (Hervorhebung von mir) verändert worden, um die Produktion durch die Körperzellen zu optimieren. Der Impfstoff mit der Bezeichnung BNT162b2.9 war gerade erst an Mäusen getestet worden, und Blutproben der Nager waren an Muik geliefert worden. Die Ergebnisse waren eindeutig: Er hatte eine weitaus bessere Antikörperreaktion ausgelöst als das sehr ähnliche modRNA-Konstrukt BNT162b2.8, das bereits als Kandidat für die klinische Studie ausgewählt worden war.“ (Project Lightspeed, S. 233)
Interessant ist an diesem Zitat eigentlich nur ein einziges Wort „die Nukleotidsequenz leicht verändert wurde, um die Produktion durch die Körperzellen zu optimieren.“
Diese „leichte“ Veränderung kennt man seit einiger Zeit unter #COptiGate[1].
Vom Wissensstand, der hier vorausgesetzt wird, befinden wir uns im Duden Biologie Abiturwissen beim Thema Proteinbiosynthese bei Pro- und Eukaryoten[2]. Bei meinem Schülerduden von 2004 ist das auf Seite 208f, also nichts, was Ugur nicht hätte wissen können und müssen! Dieser Fehler passierte also nicht auf Mittelstufenniveau, sondern auf Oberstufenniveau, was schon ein gewisser Fortschritt ist. Man freut sich ja mittlerweile über jede kleine Anhebung des Niveaus, auf dem man argumentieren kann, 9. Klasse Immunbiologie wurde mit mittlerweile schon ein wenig langweilig. Auf zum Abitur!
Die DNA setzt sich aus den vier Nukleoiden A, T, G, C zusammen. A paart mit T und G paart mit C in einem DNA-Doppelstrang. Je 3 Nukleoide ergeben ein sogenanntes Triplet. Je nach Tripletzusammensetzung werden unterschiedliche Aminosäuren oder Stopps für die Proteinfabrik Ribosom kodiert.
Abbildung 1 Schülerduden Biologie (2004) S. 203 Genetischer Code
Dekodiert werden die Triplets mittels der Transport-tRNA, die für das Ribosom übersetzt, welche Aminosäure laut mRNA Bauplan als nächstes verbaut werden soll. Transport RNA deswegen, weil jede dieser tRNA-Moleküle eine Aminosäure mit sich schleppt und quasi zum Ribosom transportiert. An dieser Stelle kommt eigentlich noch das N1-Methylpseudouridinproblem hinzu, welches aus der vielfach vermarkteten mRNA die korrekter beschriebene modifiziert (mod)RNA in der Plörre macht. Das werde ich aber der Verständlichkeit halber als eigenen Artikel ausgliedern, weil es sonst zu komplex wird.
Manche Aminosäuren haben viele verschiedene Triplets, die für sie kodieren. Manche Aminosäuren nur wenige Triplets.
In der Schule lernt man, dass jeder Tripletcode hat seine eigene dekodierende tRNA hat. Die als Decoder fungierende tRNA kommt, je nach Tripletcode, in sehr unterschiedlichen Mengen vor. Manche tRNAs sind häufig, manche sind selten. Das variiert zudem noch von Organismus zu Organismus, was es nicht einfacher macht und daher bei einem Wechsel in einen anderen Organismus beachtet werden muss, sonst kann entweder das gewünschte Protein nicht gebildet werden oder im schlimmsten Fall bringt man diesen unter Umständen aus Versehen um. Das ist Grundwissen im genetischen Labor. Immer schön den Tripletcode an den produzierenden Organismus anpassen.
Leider ist das eine SEHR vereinfachte und leider auch falsche Darstellung.
Die Realität ist wieder einmal deutlich komplexer.
Nicht jedes Codon hat überhaupt eine tRNA. Es gibt Codons, die haben gar keine eigene tRNA und werden von anderen tRNAs mit bedient. Das variiert auch nocht von Organismus zu Organismus und von Organ zu Organ.
Rot markiert die Codons, die keine eigene tRNA haben und nur über sogenannte wobbel tRNA (also an einer Stelle rumwackelnde tRNA bedient) werden, andere Codons von tRNAs wo sowohl als auch zutrifft. Was passiert wohl, wenn da ein N1-Methylpseudouridin verbaut ist?!
(A critical analysis of codon optimization in human therapeutics - PubMed (nih.gov))
Nicht zum Grundwissen gehört des Weiteren, dass es zudem wohl deutlich mehr tRNAs gibt, als wir bisher dachten und diese wohl auch eine regulatorische Funktion haben könnten, die wir noch nicht verstehen[3]. Es handelt sich um die sogenannten tRNA Isodecoder über deren Funktion spekuliert wird.
„Das Überwiegen der tRNA-Isodecoder - die mehr als die Hälfte aller in höheren Säugetieren beobachteten tRNA-Gene ausmachen - war jedoch unerwartet und ihre funktionelle Bedeutung ist noch nicht geklärt“ (However, the preponderance of tRNA isodecoders – making up more than half of all tRNA genes observed in higher mammals – was unexpected and its functional importance is not yet understood.)[4]
Unerwartet, wer konnte es nur ahnen, dass da etwas unerwartet sein könnte? Wie sich diese Isodekoder wohl bei N1-Methylpseudouridin verhalten? Aber das ist ja ein anderes Thema.
Richtig spannend wird es, wenn man bedacht hätte, was Ugur – immerhin ordentlicher Professor und Arbeitsgruppenleiter am Institut für Immunologie der Universität Mainz- aus unerklärlichen Gründen nicht getan hat, dass tRNAs möglicherweise auch noch gewebespezifisch in unterschiedlichen Konzentrationen vorhanden sein könnten, wie man das von der Seidenraupe seit 1979 weiß[5]. Hat man für den Menschen leider nicht so genau untersucht, also gar nicht. Man konnte aber beobachten, dass das Spike-Proteine, je nach Zellkultur (also ein gewisser Hinweis auf eine mögliche Organspezifität) in unterschiedlicher Menge an der Zelloberfläche exprimiert wird, während andere Zelllinien wiederum das Spike-Protein bevorzugt als freies Spike-Protein freisetzen (sezernieren)[6] [7]. Die Variation kann natürlich sowohl von den komplett unterschiedlichen Organen stammen, von denen die Zellkulturen abgeleitet sind als auch, dass es sich um Zellkulturen von unterschiedlichen Menschen handelt. Aber auch das, wäre ein Warnsignal, dass wir nichts verstanden haben, nicht ansatzweise, was bei der Proteinproduktion in den verschiedenen Zelltypen passiert.
Was hat Ugur gemacht? Er will von den Körperzellen schnell und viel Protein herstellen lassen und arbeitet ERNEUT nach dem Motto viel hilft viel. «Da wir nicht wissen, was nötig ist, sollten wir beim Maximum anfangen», hatte Ugur erklärt.“ (Projekt Lightspeed, S. 220).
So einfach, wie es im Biologiebuch steht ist es dann wohl nicht in der biologischen Realität. Aber selbst das Schulbuchwissen hätte eigentlich schon als Warnung reichen müssen, würde man annehmen.
Hier müssen wir auf das Thema Proteinfaltung eingehen: am Ribosom entsteht zuerst einmal nur eine lange Aneinanderreihung von Aminosäuren – abhängig von der Sequenzen und den Triplets der TRNAs werden diese aneinander gebastelt. Dann aber muss sich das eigentliche funktionelle Protein bilden – manche werden zu kugeln, andere zu langen fasern usw. das ist ein sehr komplexer Vorgang, fast wie Origami. Und wie bei Origami beeinflusst die Sorgfalt und Geschwindigkeit mit der gefaltet wird, auch wie genau hinterher alles ordentlich da landet, wo es soll und der Kopf des Kranichs landet a auch wirklich an der Kopfposition und nicht am Hintern, bzw. dass man überhaupt erkennt, was es eigentlich einmal werden sollte.
Und das, ist nur ein gaaaaaaaaaaaaanz einfaches Origami. Es gibt es noch ganz andere Kaliber wie Origami Samurais:
Genauso unterschiedlich schwierig wie ein Kranich im Vergleich zu einem Origami Samurai ist es auch bei den Proteinen mit dem Falten. Es gibt kleine Kranich-Proteine, die schnell und spontan falten und dann eben Samurai-Proteine. Die sind ein klein wenig komplexer. Einen Origamikranich kann man mit Kindern falten, einen Samurai eher nicht. Das Spike Protein ist ein Samurai.
Abbildung 2 Schülerduden Biologie (2004) S. 210 Translation und das allosterische Dreistellenmodell
Hat man einen Tripletcode verbaut, der eine seltene tRNA zur Dekodierung braucht, wird die Produktion des Eiweißes langsamer, weil der Nachschub an tRNA nicht so schnell fließt[8]. Das ist wie beim Legobasteln: die häufigen Bausteine kann man viel schneller zusammenstecken, also wenn man in der Wühlkiste ewig nach dem einen seltenen Teil suchen muss.
Das beeinflusst MASSIV die Faltung eines Eiweißes und ist von der Natur extra so konzipiert, damit ein Eiweiß an kniffligen Stellen Zeit hat, richtig zu falten.
Oder um aus dem Waudby Review zu zitieren: „Erstens wird die Translationsrate durch die Zeit für das Auffinden und die Dekodierung verwandter tRNA-Moleküle begrenzt, die wiederum von den jeweiligen tRNA-Konzentrationen sowie von der Konkurrenz mit verwandten tRNAs und synonymen Mutationen zwischen reichlich vorhandenen und seltenen tRNAs abhängt; es wurde beobachtet, dass diese Veränderungen die Translationsrate um Größenordnungen beeinflussen.“
Man lasse sich das Wort um GRÖSSENORDNUNGEN[9] [10] mal auf der Zunge zergehen.
Abbildung 3 Schülerduden Biologie (2004) S. 211 Regulation der Genaktivität
Wenn man sich das Bild aus dem Schülerduden mal genau anschaut, steht da bereits etwas von langsam? Schnell? Genau das Problem, das ich bereits beschrieben habe, oder um es wissenschaftlich niveauvoller zu belegen, als mit einem alten Schülerduden aus dem Jahr 2004:
Es gibt aktuell eine beliebte Theorie, wie Protein falten: Die Energielandschaft. Entlang dieser Energielandschaft faltet das Protein energetisch bergab und sucht sich so seinen Weg ins energetische Tal der optimalen Faltung. Der Weg hinab ins Tal wird räumlich dadurch eingeschränkt, dass das Protein bereits zu falten beginnt, während es aus dem Ribosom hinten herauskommt. Da ein Teil der Aminosäurekette noch im Ribosom steckt, bzw. noch gar nicht hergestellt wurde, kann nur das falten, was hinten aus dem Ribosom baumelt. Das Ribosom selbst hilft wohl auch noch bei der Faltung mit, mit irgendwelchen äußeren Strukturen (die man, Überraschung!, noch nicht verstanden hat). Da es also prinzipiell schon mal einige statisch, räumliche Einschränkungen bei der Faltung gibt, kann das Protein bei der Faltung nicht die kompletten Möglichkeiten dieser Energielandschaft nutzen[11].
Das klingt auf den ersten Blick schön gelehrt und als wenn man Ahnung hätte von was man da redet. Schaut man ins Detail, stellt man schnell fest, dass einem diese wunderbare Theorie der Energielandschaft nichts bringt, weil sie einem nicht ansatzweise erklären kann, welche Aminosäure oder Struktur des Proteins wann welche Form an welcher Stelle im Raum einnimmt oder einnehmen wird. Gelehrte heiße Luft, die verbergen soll, dass man überhaupt keine Ahnung hat, wie und warum das Protein zu diesem Zeitpunkt gerade so faltet, wie es faltet und nicht anders. Man kann aber trefflich über diese Theorie philosophieren und in einem ist man sich letztendlich einig: Ja, die Gesamtenergie wird im Endprodukt wohl am niedrigsten sein. Alla gut! Würden die Lallers (alte SWR2 Radio Comedy) an dieser Stelle wohl sagen.
Das Review von Waudby hat dann aber wunderschön ausformuliert, was im Schülerduden nur im Bild angedeutet ist:
Wenn die Translation (Übersetzung der mRNA in Protein) schnell ist (kTranslation > kFaltung), dann wird die Faltung von einem ungeordneten Zustand in der Polypeptidkette in voller Länge eingeleitet, und das Risiko der Bildung von kinetisch gefangenen und potenziell fehlgefalteten Zwischenstufen ist erhöht (magentafarbene Faltungsweg). […] Wenn jedoch die Faltung im Verhältnis zur Translation schnell ist (kFaltung > kTranslation), dann ist es unwahrscheinlich, dass dieser fehlgefaltete Zustand auftritt, da die meisten Polypeptide sich sequenziell über die N-terminale Domäne falten (orangefarbener Faltungsweg). Daher ist es in dieser Situation eindeutig wünschenswert, dass die Translationsrate im Verhältnis zur Faltungsrate reduziert wird, damit der co-translationale Faltungsprozess die effiziente Synthese vollständig gefalteter Proteine möglichst effektiv unterstützen kann.
Im Klartext, wenn zu schnell zu viel abgelesen wird, steigt die Wahrscheinlichkeit er Proteinfehlfaltung und das weiß man auch. (Es kann natürlich in Einzelfällen wünschenswert sein, dass empfindliche Bereiche eines Proteins schneller produziert werden und schneller falten, damit sie keinen Schaden nehmen. Daher wird die Geschwindigkeit der Proteinproduktion über die jeweilige Verfügbarkeit der tRNAs reguliert.)
Zur Erinnerung: Was hat Ugur gemacht? Er will von den Körperzellen schnell und viel Protein herstellen lassen und arbeitet ERNEUT nach dem Motto viel hilft viel. «Da wir nicht wissen, was nötig ist, sollten wir beim Maximum anfangen», hatte Ugur erklärt.“ (Projekt Lightspeed, S. 220).
Da gibt es noch eine „Kleinigkeit“ die Ugur nicht bedacht hat: Die Evolution! Jahrtausende, wenn nicht Jahrmillionen Jahre, in denen sich Sequenzen über Versuch und Irrtum aus gutem Grund zu einer ganz bestimmten Codonnutzung (codon usage) optimiert haben.
Waudby schreibt es in seinem Review in ganz einfach zu verstehenden Worten, die man auch Fachfremder beim Querlesen verstehen kann, wenn man denn will:
Es gibt inzwischen eindeutige Belege dafür, dass kotranslationale Faltung und Translationskinetik einer evolutionären Selektion unterliegen[12] [13] , und dass eine Störung dieser Abstimmung zu Fehlfaltung und beeinträchtigter Proteinsynthese führen kann[14] [15] [16] [17].
Ach nee, was Du nicht sagst Sherlock.
Schon, wenn man in den im obigen Satz zitierten Papern nur den Abstract liest, hätte einem klar sein müssen, dass Codon Optimierung eine echt so richtig schlechte Idee ist, so richtig, richtig saudumm:
Die Wahl der Codons kann die lokale Translationskinetik während der Proteinsynthese beeinflussen.[18]
Wir beschreiben einen robusten statistischen Ansatz zur Identifizierung von Loci innerhalb von Genen, die sowohl signifikant mit langsam übersetzten Codons angereichert als auch evolutionär konserviert sind.[19] – Hat man das vorher überprüft bei der Codon Optimierung?
Teilweise steht es sogar direkt in der Überschrift der Publikationen wie:
Nicht optimaler Codongebrauch beeinträchtigt Expression, Struktur und Funktion des Uhrenproteins FRQ[20], da muss man nicht einmal das ganze Paper oder auch nur den Abstract lesen.
So richtig, richtig lustig, wird es, wenn eine stille Mutation, welche die Aminsäuresequenz des Proteins NICHT verändert hat zu einer komplett anderen Substratspezifiät führt[21]. Oups…
„Wir stellen die Hypothese auf, dass das Vorhandensein eines seltenen Codons, das durch den synonymen Polymorphismus gekennzeichnet ist, den Zeitpunkt der kotranslationalen Faltung und des Einbaus von P-gp in die Membran beeinflusst und dadurch die Struktur der Substrat- und Inhibitor-Interaktionsstellen verändert.“
Zu dem Themenkomplex gibt es direkt auch ein ganzes Paper:
„In allen Genomen werden die meisten Aminosäuren durch mehr als ein Codon kodiert. Synonyme Codons können die Proteinproduktion und -faltung beeinflussen, aber der Mechanismus, der die Codonverwendung mit der Proteinhomöostase verbindet, ist nicht bekannt.“[22]
Ich denke, dieser Satz aus dem Waudby Review sagt alles, dem ist nichts hinzuzufügen:
Die Geschwindigkeit der Translation im Verhältnis zur Faltungsgeschwindigkeit ist von zentraler Bedeutung für den Ausgang und das Ergebnis der co-translationalen Faltung. (The rate of translation relative to that of folding is of central importance in defining the outcome of co-translational folding).
Und an dieser Geschwindigkeit hat Ugur komplett sinnbefreit herumgeschraubt und geglaubt, dass schon nichts passieren würde. 6! Setzen!
Sind entsprechende Folgen der in den Publikationen angedeuteten Codonoptimierung für das Spike-Protein mittlerweile nachgewiesen? Ja, sogar von Ugur selbst, man kann es kaum glauben:
„Die Nukleotidsequenz für den knubbeligen Stachel war leicht (Hervorhebung von mir) verändert worden, um die Produktion durch die Körperzellen zu optimieren. Der Impfstoff mit der Bezeichnung BNT162b2.9 war gerade erst an Mäusen getestet worden, und Blutproben der Nager waren an Muik geliefert worden. Die Ergebnisse waren eindeutig: Er hatte eine weitaus bessere Antikörperreaktion ausgelöst als das sehr ähnliche modRNA-Konstrukt BNT162b2.8, das bereits als Kandidat für die klinische Studie ausgewählt worden war.“ (Project Lightspeed, S. 233)
Ugur Sahin schreibt in seinem Buch selbst, dass das BNT162b2.9 immunogener ist als das Konstrukt BNT162b2.8, welches in der Phase 1 und 2 getestet wurde und das, obwohl es sich „nur“ um eine Codonoptimierung handelt[23], die, wie wir bereits beschrieben haben, zu einem komplett anderen Protein mit vollkommen anderen Eigenschaften führen kann, obwohl die Aminosäuresequenz identisch ist, rein durch einen anderen Faltungsweg aufgrund einer einzigen stillen Mutation bzw. sehr, sehr vieler stiller Mutationen im Falle der Codonoptimierung.
Warum das so ist, weiß aber keiner. Die Warnzeichen hat wohl keiner verstanden, schon gar nicht Ugur. Wie auch, dazu hätte er was von Proteinfaltung oder protein engineering verstehen müssen, was niemand auf diesem Planeten so wirklich tut. Im besten Fall hat man verstanden, dass man praktisch nichts weiß und versteht, dass Proteinfaltungspaper, neben ersten harten Fakten, zum Großteil aus sehr viel heißer Luft bestehen, man das Thema daher lieber in der praktischen Anwendung extrem vorsichtig angehen sollte.
Es gibt ein interessantes australisches Pfizer Dokument[24]. In diesem Dokument findet man noch einige Perlen. So findet man auf Seite 19 des foi-2389-06.pdf den Unterschied zwischen BNT162B2.8 und BNT162B2.9.
Diese beiden Varianten haben die gleiche Aminosäuresequenz, sollten also theoretisch das identische Protein ergeben. ABER sie haben eine unterschiedliche Codon Optimierung. BioNTech hat die Codons so optimiert, dass die Antigenproduktion optimiert wurde. Ein keiner Dreh an den Sequenzen und schon wurde im selben System mehr Antigen gebildet
Nur… warum ist BNT62B2.9 immunogener als BNT162B2.8? Wie kann das sein, wenn doch angeblich das gleiche Protein dabei herauskommt. Mehr Antigen bedeutet nicht zwangsläufig mehr Antikörper, zumindest hat das keiner bei BioNTech oder Pfizer jemals belegt. Warum also reagiert der Körper heftiger auf BNT162B2.9 als auf BNT162B2.8?
Der australische Senator Rennick[25]. fragt also zu Recht im australischen Parlament: „Warum haben Sie diesen Absatz im ursprünglichen FOIA Dokument geschwärzt, wenn man berücksichtig, dass dieser beweist, dass der Impfstoff mit gain-of-funktion optimiert wurde, welche die Toxizität des Impfstoffs durch die Erhöhung der Menge des Spike-Proteins erhöhte? […] Dieser Impfstoff erhöht die Antigenexpression, richtig, des Spike-Proteins. Er erhöht die Menge des Proteins, das der Impfstoff im Vergleich zum Virus bildet. Ein herkömmlicher Impfstoff ist im Allgemeinen abgeschwächt und nicht mit einer höheren Expressionsfunktion ausgestattet. Akzeptieren Sie also, dass der Impfstoff eine Funktionserweiterung darstellt?“
Worin liegt letztendlich der Unterschied zwischen dem Protein, das produziert würde mit der Originalen Codon Sequenz, und den beiden verschiedenen Optimierungen BNT162B2.8 und BNT162B2.9? Kann es sich vielleicht letztendlich aufgrund von unterschiedlichen Faltungswegen sogar um 3 unterschiedliche Proteine mit teils sogar unterschiedlicher Funktion oder Spezifitäten handeln? Theoretisch, anhand der bekannten, etablierten Literatur, wäre das durchaus möglich. Es kommt jedenfalls in Zellkultur nicht nur ein sauberes, eindeutiges Protein heraus, sondern verschiedene Varianten[26]. Ohne entsprechende Datenlage kann man darüber am Lagerfeuer nun über Jahre hinweg genüsslich philosophieren, ohne je zu einem Ergebnis zu kommen. Theoretisch kann man mit dieser Fragestellung das alte, ritualisierte Kennenlernritual auf Konferenzen ersetzen. Aus „was wird die Menschheit ausrotten, eher ein Virus oder ein Bakterium“ könnte man seine ritualisierten Diskussionen nun zu „sind das natürliche Spike BNT162B2.8 und 9 nun das gleiche Protein, oder 2 oder 3 komplett unterschiedliche Proteine“. Man kann so abchecken, wie das Gegenüber tickt und argumentiert. Die Viren versus Bakterien Frage ist irgendwie verbrannt und problematisch, wenn man auf jemanden von Next Level treffen sollte. Dann kann es ungemütlich werden und die Party ist geplatzt.
Da keiner sich bisher die Mühe gemacht hat, diese Protein Proteinbiochemisch auch nur ansatzweise und grundlegend zu charakterisieren – oder wenn doch, dann sind die Daten im schwarzen Archiv von Ugurs Hexenküche seeehr tief vergraben, genauso tief wie in den Geheimarchiven des Vatikan, ist bisher schwer bis gar nicht zu sagen, wie sich diese potentiell unterschiedlichen Proteine verhalten könnten, angefangen bei Thermostabilität über Bindungsaffinitäten zu Bindung an andere Rezeptoren.
Mittlerweile wurde zumindest für die S1 Untereinheit des Spikes (die RBD- Rezeptor binding Domain) bewiesen, dass sie fehlfaltet[27] [28] und nicht nur das, es werden teilweise komplett andere Peptide (kleine Proteinfragmente) hergestellt die so gar nichts mit dem originalen Spike-Protein gemein haben.
Nachdem wir die Theorie der Proteinfaltung im Zusammenhang mit Codonoptimierung nun soweit erklärt haben, schauen wir uns doch mal im Detail an (soweit bekannt), was BioNTech/Pfizer da dilettantisch zusammengebraut haben. Wäre das ein Masterprojekt gewesen, wäre der Student bei mir aufgrund der missachteten, bekannten biologischen Grundlagen krachend durchgefallen.
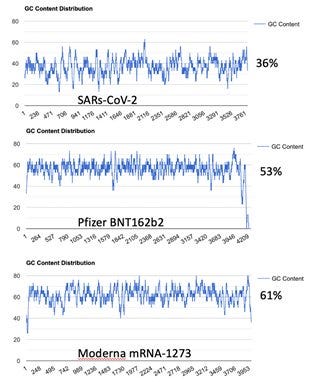
Schauen wird doch mal, wie extrem am GC Gehalt und somit an den Codons geschraubt wurde. Zur Erinnerung, ein Virus oder Pathogen ist normalerweise evolutionär an seinen Wirtsorganismus und somit an dessen tRNAs und Ribosome angepasst und hat daher aus gutem Grund eine bestimmte Codonnutzung für seine Proteine.
Kevin McKernan[29] hat sich dieses Problem mal im Detail angeschaut. Wie häufig wurden welche Basen (also A, T, C, G) verwendet.
17% höherer GC-Gehalt bei BioNTech/Pfizer und 25% mehr bei Moderna als in der Original-Spike Sequenz aus Wuhan. Das nenne ich mal sportlich, wenn man sich zurückerinnert, dass eine einzige stille Mutation bereits die Substratspezifität eines Enzyms verändern kann. Besonders mutig, wenn man so etwas dann zur Anwendung im Menschen bringt. Was kann da schon schiefgehen außer so ziemlich alles, was man sich auch nur in seinen schlimmsten Albträumen ausmalen kann.
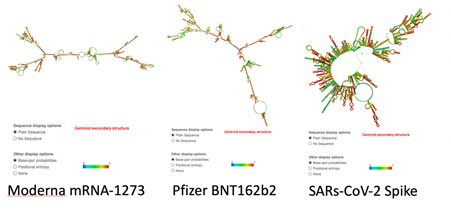
Auch lustig, dass sich dadurch nicht nur das abgelesene Protein sondern auch schon die Sekundärstruktur der mRNA/modRNA ändert. mRNA kann regulatorische Funktion haben bei der Translation, weil diese Strukturen nicht so einfach durch das Ribosom gehen, als wenn es eine gerade ungefaltete mRNA wäre.
Auch da hat Kevin McKernan genauer drauf geschaut, wie sich denn die Sekundärstruktur der mRNA (eigentlich modRNA, aber wir lassen hier erneut diese N1-Methylpseudouridinproblematik außen vor) verändert hat. Etwas, was Ugur wohl auch nicht auf dem Schirm hatte, denn wer hätte auch ahnen können, dass die 3D Struktur der mRNA möglicherweise Einfluss auf die Translationsgeschwindigkeit nehmen könnte? Dazu hätte man ja mal ein Proteinfaltungspaper wie das von Waudby lesen müssen. Der hat dazu ein komplettes Kapitel in seinem Review:
„Als Folge der Degeneration des genetischen Codes können mRNA-Sequenzen Informationen enthalten, die über die primäre Polypeptidsequenz hinausgehen, und es hat sich gezeigt, dass diese Fähigkeit die Translationsgeschwindigkeit durch mehrere sich nicht gegenseitig ausschließende Mechanismen beeinflusst.[…] (W)enn die Translationskinetik stark gestört ist, kann dies zu einer Störung der Proteostase und zu Krankheiten führen.“
Und hier wird es nun noch mal spannend:
„Zweitens können stabile mRNA-Sekundärstrukturelemente in einer begrenzten Anzahl von Fällen auch die Translationsrate verlangsamen. In vitro ist der Einfluss der mRNA-Struktur auf die Translation eindeutig und wurde mit Hilfe von Einzelmolekülmessungen im Detail verfolgt.“
Dass RNA solche Strukturen macht, lernt man normalerweise in der Vorlesung „Einführung in die Genetik“ im ersten Semester und das schon vor 20 Jahren.
So auf den ersten Blick kommen mir die codonoptimierten modRNAs schon ein wenig einfach gestrickt vor in ihrer Sekundärstruktur im Vergleich zum Original.
=== Fazit ===
Wenn ich Ugur vor Gericht gegenüberstünde, würde ich ihm bezüglich der Codonoptimierung gerne ein paar Fragen stellen:
1. Es ist aus der Literatur hinlänglich bekannt, dass selbst eine stille Mutation bereits die Faltung eines Protein so verändern kann, dass es eine andere Substratspezifität aufweist. Haben Sie den Einfluss der optimierten Codons auf die Proteintranslation und deren Einfluss auf die Proteinfaltung hin überprüft? Wenn ja, mit welchem Ergebnis? Wenn nein, warum nicht und sind sie sich des damit einhergehenden Risikos bewusst gewesen?
2. Haben Sie untersucht, warum BNT162B2.9 immunogener ist als BNT162B2.8? Wenn ja, was war das Ergebnis. Wenn nein, warum nicht?
3. Haben Sie den Unterschied der veränderten mRNA Struktur auf das Translations- und somit Faltungsverhalten des resultierenden Proteins untersucht? Wenn ja, was war das Ergebnis? Wenn nein, warum nicht?
4. Haben sie die potentiell unterschiedliche tRNA Ausstattung verschiedener menschlicher Gewebe bei der Translation des Spike-Proteins berücksichtigt?
5. Hatten Sie überhaupt jemanden im Team, der von so etwas auch nur ansatzweise eine Ahnung von protein engineering hatte oder hat?
[1] https://pingthread.com/hashtag/COptiGate
[2] Eiweißsynthese in Biologie | Schülerlexikon | Lernhelfer. (n.d.). https://www.lernhelfer.de/schuelerlexikon/biologie/artikel/eiweisssynthese
[3] Goodenbour JM, Pan T. Diversity of tRNA genes in eukaryotes. Nucleic Acids Res. 2006;34(21):6137-46. doi: 10.1093/nar/gkl725. Epub 2006 Nov 6. PMID: 17088292; PMCID: PMC1693877. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1693877/
[4] Hughes LA, Rudler DL, Siira SJ, McCubbin T, Raven SA, Browne JM, Ermer JA, Rientjes J, Rodger J, Marcellin E, Rackham O, Filipovska A. Copy number variation in tRNA isodecoder genes impairs mammalian development and balanced translation. Nat Commun. 2023 Apr 18;14(1):2210. doi: 10.1038/s41467-023-37843-9. PMID: 37072429; PMCID: PMC10113395. https://pubmed.ncbi.nlm.nih.gov/37072429/
[5] Hagenbüchle O, Larson D, Hall GI, Sprague KU. The primary transcription product of a silkworm alanine tRNA gene: identification of in vitro sites of initiation, termination and processing. Cell. 1979 Dec;18(4):1217-29. doi: 10.1016/0092-8674(79)90234-4. PMID: 519766. https://pubmed.ncbi.nlm.nih.gov/519766/
[6] Joomi. (2023, May 17). New study shows how little we know about how mRNA vaccines “work.” Substack. https://joomi.substack.com/p/new-study-shows-how-little-we-know
[7] Cari L, Naghavi Alhosseini M, Mencacci A, Migliorati G, Nocentini G. Differences in the Expression Levels of SARS-CoV-2 Spike Protein in Cells Treated with mRNA-Based COVID-19 Vaccines: A Study on Vaccines from the Real World. Vaccines (Basel). 2023 Apr 21;11(4):879. doi: 10.3390/vaccines11040879. PMID: 37112792; PMCID: PMC10144021. https://pubmed.ncbi.nlm.nih.gov/37112792/
[8] Zhang G, Hubalewska M, Ignatova Z. Transient ribosomal attenuation coordinates protein synthesis and co-translational folding. Nat Struct Mol Biol. 2009 Mar;16(3):274-80. doi: 10.1038/nsmb.1554. Epub 2009 Feb 8. PMID: 19198590. https://pubmed.ncbi.nlm.nih.gov/19198590/
[9] Sørensen MA, Kurland CG, Pedersen S. Codon usage determines translation rate in Escherichia coli. J Mol Biol. 1989 May 20;207(2):365-77. doi: 10.1016/0022-2836(89)90260-x. PMID: 2474074. https://pubmed.ncbi.nlm.nih.gov/2474074/
[10] Fluitt A, Pienaar E, Viljoen H. Ribosome kinetics and aa-tRNA competition determine rate and fidelity of peptide synthesis. Comput Biol Chem. 2007 Oct;31(5-6):335-46. doi: 10.1016/j.compbiolchem.2007.07.003. Epub 2007 Aug 15. PMID: 17897886; PMCID: PMC2727733. https://pubmed.ncbi.nlm.nih.gov/17897886/
[11] Waudby CA, Dobson CM, Christodoulou J. Nature and Regulation of Protein Folding on the Ribosome. Trends Biochem Sci. 2019 Nov;44(11):914-926. doi: 10.1016/j.tibs.2019.06.008. Epub 2019 Jul 10. PMID: 31301980; PMCID: PMC7471843. https://pubmed.ncbi.nlm.nih.gov/31301980/
[12] Pechmann S, Frydman J. Evolutionary conservation of codon optimality reveals hidden signatures of cotranslational folding. Nat Struct Mol Biol. 2013 Feb;20(2):237-43. doi: 10.1038/nsmb.2466. Epub 2012 Dec 23. PMID: 23262490; PMCID: PMC3565066. https://pubmed.ncbi.nlm.nih.gov/23262490/
[13] Jacobs WM, Shakhnovich EI. Evidence of evolutionary selection for cotranslational folding. Proc Natl Acad Sci U S A. 2017 Oct 24;114(43):11434-11439. doi: 10.1073/pnas.1705772114. Epub 2017 Oct 10. PMID: 29073068; PMCID: PMC5664504. https://pubmed.ncbi.nlm.nih.gov/29073068/
[14] Zhang G, Hubalewska M, Ignatova Z. Transient ribosomal attenuation coordinates protein synthesis and co-translational folding. Nat Struct Mol Biol. 2009 Mar;16(3):274-80. doi: 10.1038/nsmb.1554. Epub 2009 Feb 8. PMID: 19198590. https://pubmed.ncbi.nlm.nih.gov/19198590/
[15] Zhou M, Guo J, Cha J, Chae M, Chen S, Barral JM, Sachs MS, Liu Y. Non-optimal codon usage affects expression, structure and function of clock protein FRQ. Nature. 2013 Mar 7;495(7439):111-5. doi: 10.1038/nature11833. Epub 2013 Feb 17. PMID: 23417067; PMCID: PMC3629845. https://pubmed.ncbi.nlm.nih.gov/23417067/
[16] Kimchi-Sarfaty C, Oh JM, Kim IW, Sauna ZE, Calcagno AM, Ambudkar SV, Gottesman MM. A "silent" polymorphism in the MDR1 gene changes substrate specificity. Science. 2007 Jan 26;315(5811):525-8. doi: 10.1126/science.1135308. Epub 2006 Dec 21. Erratum in: Science. 2007 Nov 30;318(5855):1382-3. Erratum in: Science. 2011 oCT 7;334(6052):39. PMID: 17185560. https://pubmed.ncbi.nlm.nih.gov/17185560/
[17] Kim SJ, Yoon JS, Shishido H, Yang Z, Rooney LA, Barral JM, Skach WR. Protein folding. Translational tuning optimizes nascent protein folding in cells. Science. 2015 Apr 24;348(6233):444-8. doi: 10.1126/science.aaa3974. PMID: 25908822 https://pubmed.ncbi.nlm.nih.gov/25908822/
[18] Pechmann S, Frydman J. Evolutionary conservation of codon optimality reveals hidden signatures of cotranslational folding. Nat Struct Mol Biol. 2013 Feb;20(2):237-43. doi: 10.1038/nsmb.2466. Epub 2012 Dec 23. PMID: 23262490; PMCID: PMC3565066. https://pubmed.ncbi.nlm.nih.gov/23262490/
[19] Jacobs WM, Shakhnovich EI. Evidence of evolutionary selection for cotranslational folding. Proc Natl Acad Sci U S A. 2017 Oct 24;114(43):11434-11439. doi: 10.1073/pnas.1705772114. Epub 2017 Oct 10. PMID: 29073068; PMCID: PMC5664504. https://pubmed.ncbi.nlm.nih.gov/29073068/
[20] Zhou M, Guo J, Cha J, Chae M, Chen S, Barral JM, Sachs MS, Liu Y. Non-optimal codon usage affects expression, structure and function of clock protein FRQ. Nature. 2013 Mar 7;495(7439):111-5. doi: 10.1038/nature11833. Epub 2013 Feb 17. PMID: 23417067; PMCID: PMC3629845. https://pubmed.ncbi.nlm.nih.gov/23417067/
[21] Kimchi-Sarfaty C, Oh JM, Kim IW, Sauna ZE, Calcagno AM, Ambudkar SV, Gottesman MM. A "silent" polymorphism in the MDR1 gene changes substrate specificity. Science. 2007 Jan 26;315(5811):525-8. doi: 10.1126/science.1135308. Epub 2006 Dec 21. Erratum in: Science. 2007 Nov 30;318(5855):1382-3. Erratum in: Science. 2011 oCT 7;334(6052):39. PMID: 17185560. https://pubmed.ncbi.nlm.nih.gov/17185560/
[22] Buhr F, Jha S, Thommen M, Mittelstaet J, Kutz F, Schwalbe H, Rodnina MV, Komar AA. Synonymous Codons Direct Cotranslational Folding toward Different Protein Conformations. Mol Cell. 2016 Feb 4;61(3):341-351. doi: 10.1016/j.molcel.2016.01.008. PMID: 26849192; PMCID: PMC4745992. https://pubmed.ncbi.nlm.nih.gov/26849192/
[23] https://phmpt.org/wp-content/uploads/2023/01/125742_S21_M1_pharmacovigilance-plan.pdf#page=1 Seite 9f
[24] https://www.tga.gov.au/sites/default/files/foi-2389-06.pdf
[25] Rennick, O. O. S. G., & Rennick, O. O. S. G. (2023, March 14). Gain-of-function research in Australia: Why the secrecy? - Senator Gerard Rennick. Senator Gerard Rennick. https://gerardrennick.com.au/gain-of-function-research-in-australia-why-the-secrecy
[26] Jiang H, Mei YF. SARS-CoV-2 Spike Impairs DNA Damage Repair and Inhibits V(D)J Recombination In Vitro. Viruses. 2021 Oct 13;13(10):2056. doi: 10.3390/v13102056. Retraction in: Viruses. 2022 May 10;14(5): PMID: 34696485; PMCID: PMC8538446. https://pubmed.ncbi.nlm.nih.gov/34696485/
[27] Patterson, B. W., Francisco, E. B., Yogendra, R., Long, E., Pise, A., Beaty, C. D., Osgood, E., Bream, J. B., Kreimer, M., Heide, R. S. V., Guevara-Coto, J., Mora, R., & Mora, J. (2022). SARS-CoV-2 S1 Protein Persistence in SARS-CoV-2 Negative Post-Vaccination Individuals with Long COVID/ PASC-Like Symptoms. Research Square (Research Square). https://doi.org/10.21203/rs.3.rs-1844677/v1
[28] J. (2022, September 29). What proteins are we actually getting from the COVID vaccines? https://joomi.substack.com/p/what-proteins-are-we-actually-getting?utm_campaign=post&utm_medium=web&utm_source=direct
[29] McKernan, K., Kyriakopoulos, A. M., & McCullough, P. A. (2021, November 25). Differences in Vaccine and SARS-CoV-2 Replication Derived mRNA: Implications for Cell Biology and Future Disease. https://doi.org/10.31219/osf.io/bcsa6
Update:
09.05.2024 (A critical analysis of codon optimization in human therapeutics - PubMed (nih.gov))










Erschreckend einfach dargelegt wie Scheiße diese neuen "Impfstoffe sind. Traurig, das ich Sie erst jetzt gefunden habe. Übrigens, wenn es um Naturwissenschaftliche Realsatire gehen sollte, hätte ich da einen für Sie... ist auch nur etwas mehr als 20 Jahre alt und auch nicht aus dem bilogischen Bereich sonder aus dem physikalischen... also da wo man ziemlich schnelle und einfach sagen kann ob eine Theorie kappes ist oder nicht. https://archive.org/details/free-fall-nist-ncstar-1-a
Und des weiteren würde ich dann den Vortrag des Dr. Ansgar Schneider empfehlen, ist zwar 2 1/2 Stunden lang (https://odysee.com/@Politik_bunter_mix:f/9_11_aus_der_Sicht_der_Physik_-_Ansgar_Schneider_ueber_Wissenschaft_und_gesellschaftliche_Leugnung:f), dürfte aber für einen Naturwissenschaftler durchaus von Interesse sein. Zumal in dem Vortrag ein kleiner Hinweis auf die Zukunft auftaucht, von dem Dr. Schneider damals nicht wusste wie wichtig das noch sein könnte. (https://archive.org/details/cost-conspiracytheories-vaccination-programmes)
Eine Frage hätte ich aber noch: Haben Sie das auch auf Englisch? Falls nicht, würde ich dringenst empfehlen dies in irgendeiner Form nachzureichen, denn die Reichweite ihrer Beiträge sollte meiner Meinung nach unbedingt vergrößert werden.
PS; Ich vermute SIehaben sich die Haare ausgerissen als Sie die Erklärungen im Fernsehen und Radio gehört haben bezüglich der Wirkungsweise der "Impfstoffe": "Der Impfstoff wird in den Arm gespritzt, verbleibt dort und Zellen nehmen die mRNA auf und trainieren dann das Immunsystem." Das dürfte noch nicht einmal für die Mittelstufe gereicht haben.
Zur weiteren Lektüre: https://ghostfromthefuture.substack.com/p/i-have-questions
https://ghostfromthefuture.substack.com/p/now-that-your-eyes-are-open
Und ich empfehlen ebenfalls noch das Interview zwischen Katie Halper, Matt Taibbi und max Blumenthal zum Thema Zensur (https://rumble.com/v3e43qr-matt-taibbi-and-max-blumenthal-on-neoliberal-censorship.html). Leider haben alle drei keine Ahnung von dem, was Sie hier der Öffentlichkeit preisgeben. Leider. Da müsste man überlegen wie all dies zusammen gebracht werden könnte. Und zwar mit Warpspeed... =;)
Übrigens: Kenne Sie den Blog von Doorless Carp? @DoorlessCarp🐭 sehr lesenswert!