Integriert die Plörre nun oder nicht?
Entspannt euch, die DNA-Fragmente der Plörre müssen nicht integrieren, um Schaden anzurichten
Die Diskussion, ob die Plörre nun in das Genom integriert oder nicht, ist ja immer noch eine heiße Frage, weil, mir zumindest, noch kein endgültiger Laborbeweis mittels Sequenzierung, der das Plasmid oder Teile davon in einem Tumor nachweist, bekannt ist.
Es gibt aber mittlerweile einiges an Daten, die ich hier mal zusammenfassen will, damit ich das nicht jedes Mal einzeln beantworten muss, wenn mich jemand anschreibt. Link setzen ist einfacher und spart mir langfristig einfach Arbeit.
Ich bin nicht allwissend, ich kenne nicht alle Paper zu diesem Thema, aber das, was ich weiß, kann ich zumindest zusammenfassen. Wer weiterführende Informationen hat, gerne in den Kommentarbereich.
Wir beginnen historisch mit Dingen, die man eigentlich hätte wissen sollen.
=== Insertionsmutagenese ===
Insertionsmutagenese (insertional mutagenesis) ist eine alte Methode der Genetik, um Gene zu lokalisieren und zu charakterisieren, bevor es die Sequenzierung ganzer Genome zum „erschwinglichen“ Preis gab.
Ja, es gab eine Zeit vor der Sequenzierung, bzw. eine Zeit, in der eine Doktorarbeit von 5 Jahren darin bestand, ein einzelnes Gen zu klonieren oder zu identifizieren oder zu sequenzieren. Ein solches Beispiel ist das Lysozym Gen, das von der AG Sippel identifiziert wurde, damals 1980[1]. Das weiß ich, weil Sippel der Chef meines Doktorvaters war und gerne von den alten Zeiten erzählte, als man noch nach Frankfurt an den Flughafen fuhr, um die Restriktionsendonucleasen, die aus USA importiert werden mussten, direkt am Flughafen aus dem Zoll zu holen. Damals konnte man noch ein Paper veröffentlich, dass „nur“ eine einfache Klonierung in ein Plasmid beschrieb[2], was heute eine Routinetechnik ist. Wir sprechen also von den Anfängen der Genetik. Soooooo alt ist das Problem, vor dem wir stehen und so bekannt hätte es daher auch Ugur sein sollen, hätte er im ersten Semester bei Professor Sippel in der Vorlesung gesessen, so wie ich.
Damit man auch wirklich verstand, was einem erzählt wurde, gab es zu dieser Vorlesung eine zweistündige Pflichtübung mit Anwesenheitskontrolle. Danach saßen die Grundlagen der Genetik. Entsprechend verhasst war diese Vorlesung und erst recht die Übungen dazu. Im Nachhinein bin ich aber sehr dankbar dafür, dass die Genetiker zu meiner Zeit so rigoros grausam zu uns Ersties waren. Davon profitiere ich aktuell immer noch, obwohl ich Genetik letztendlich nicht vertieft habe, weil die Genetiker extrem unberechenbar waren in Prüfungen.
Insertionsmutagenese funktioniert recht einfach:
1. Man sorgt dafür, dass ein Gen/DNA-Fragment in das Genom einer Zellkultur oder eines Versuchstiers integriert.
2. Dann wartet man, ob ein Gen ausgeschaltet wurde und welchen Effekt das auf das Versuchssystem hat. Entweder die Zelle entartet zu einer Krebszelle oder es können auch andere Defekte auftreten, sogenannte Knock-out Mutanten. Wenn man das mit embryonalen Stammzellen von Mäusen macht und diese dann Mäusen einpflanzt und schaut, ob die Babymäuse gesundheitliche Probleme haben, kann man sich auf die Suche nach funktionalen Genen machen, die verschiedenste Effekte haben könnten.
Manchmal hat man kein Krebsgen identifiziert, manchmal ein anderes nützliches Gen, das man charakterisieren kann. Reines Glücksspiel. In einer Promotion herzlich egal, Hauptsache man hat ein Ergebnis. Das Ergebnis kann auch sein: Wenn das Gen kaputt ist, sterben homozygote Mausbabys direkt nach der Geburt bzw. haben folgende Organschäden…
3. Man schaut anschließen, wo das Konstrukt, das man zum kaputtmachen des Gens verwendet hat, abgeblieben ist. Denn man weiß normalerweise, was man als Konstrukt zu diesem Zwecke eingesetzt hat, und muss nun nur mit den üblichen Methoden nachschauen wo sich dieses Konstrukt im Genom gerade herumtreibt.
DAS ist historisches Wissen aus den 1980er und 1990er Jahren, welches man eigentlich in der Vorlesung „Einführung in die Genetik“ im ersten Semester gelernt haben sollte.
Diese Methoden werden in verfeinerter Form auch heute noch in der Identifizierung von Tumorsuppressorgenen oder Onkogenen eingesetzt[3], wenn auch ausgefeilter und verfeinerter als vor 40 Jahren.
Die Grundlage ist dieselbe: Zerstöre zufällig ein Gen, indem du irgendeinen DNA-Schrott mitten rein oder kurz davor klonierst und analysiere den Effekt, wenn es denn einen gibt.
=== Was kann denn so ins Genom integrieren ===
In der kontrollierten Forschung, in welcher man Insertionsmutagenese einsetzt, werden etablierte, kontrollierte Methoden eingesetzt, die ein nachvollziehbares Ergebnis erzeugen. Man verwendet als zerstörerische Elemente z. Bsp.
· Transposons (springende Gene)
· Retroviren
· Lentiviren
Was auffällt, hier werden zwei Viren verwendet, um Krebs oder andere Gendefekte zu erzeugen, indem diese zufällig ins Genom integrieren. Etablierte Standardmethoden, wie schon erwähnt.
Was hat man für die Plörre von J&J oder AZ verwendet?
Adenoviren?
Wozu hat man Adenoviren 1999 verwendet?
Gentherapie?
Ich sage nur: Jesse Gelsinger[4].
Ich gehe davon aus, wenn man ein Virus für Gentherapie einsetzt, dann will man, dass es stabil ins Genom integriert, warum sonst sollte man dieses Virus sonst für Gentherapie einsetzen. Komplett blöd war man 1999 nicht, dass man dafür Adenoviren genommen hat[5] [6] [7] [8].
Man kann also davon ausgehen, dass die Menschen, die J&J (AD 26) oder AZ (ChAdOx1) Plörre bekommen haben, die beide auf Adenoviren basieren, nun ein erhöhtes Krebsrisiko haben dürften, weil diese ihre Payload zufällig ins Genom integrieren wie man das von Insertionsmutagenese seit 40 Jahren kennt. Das mag bei Tumoren egal sein, die sind ohnehin entartet, bei gesunden Zellen jedoch könnte sich das als dezent kontraproduktiv erweisen.
Was mir persönlich unangenehm auffällt ist, dass die Verbindung zwischen Insertionsmutagenese, Adenovirengentherapie/Impfung und Krebs von den Forschern nicht hergestellt wird. Es scheint fast so, als wenn der Glaube daran, Krebs durch Viren heilen zu können/wollen, den Forschern das Hirn vernebelt und sie nicht erkennen wollen, dass sie damit Krebs in gesunden Zellen erzeugen können. Aber vielleicht liege ich ja einfach falsch und bin eine böse Ungläubige.
=== Welche Möglichkeiten der Insertion hat die modRNA Plörre? ===
Es wurde lange spekuliert, dass die modRNA Plörre über Line-1 integrieren könnte. Also, dass das Enzym Line-1 die modRNA in DNA übersetzt und diese dann integriert. Lange glaubte man auch einen entsprechenden in vitro Beweis dafür zu haben, zumindest in Leberzellen[9]. Der Haken an der Geschichte… die Forscher haben nicht nachgeschaut, ob die Plörre mit DNA verunreinigt ist und was da wirklich integriert hat. Damit ist dieses Paper, meiner Meinung nach, für den Arsch, seit Kevin McKernen bewiesen hat, dass sowohl Plasmide als auch DNA-Fragmente in der Plörre enthalten sind[10] [11] [12] [13]. Seit den ersten Substacks von Kevin aka Anandamide, sind diese Daten von diversen Laboren bestätigt worden[14] [15] [16] auch für Deutschland[17] durch das MMD Labor von Professor König. Somit sind nun endlich auch Daten für die eigentlich komplett getrennte Lieferkette, die nur Deutschland und die Türkei betrifft, vorhanden, bisher leider ohne Sequenz, um zu verifizieren, ob das gleiche Plasmid mit SV40 Promotor verwendet wurde.
DAS ist aber kein Beweis, dass das Plasmid selbst bzw. die DNA -Fragmente auch tatsächlich stabil in den Zellkern intergiert haben! Mir ist bisher kein einziges Paper oder kein einziger Laborbeweis bekannt, in welchem nachgeschaut worden wäre ob Fragmente oder Plasmid der Plörre in der DNA von Tumoren nachweisbar wären. Und das hat auch einen Grund.
Es gibt aber neben Line-1 noch einige weitere reverse Transkriptasen, die man erst entdeckt hat. Was die mit der modRNA machen kann man nicht ansatzweise vorhersagen:
2017: Ribozyme sind RNA-Moleküle mit katalytischer Aktivität (https://flexikon.doccheck.com/de/Ribozym). Es gibt wohl auch welche, die als reverse Transkriptase funktionieren. A reverse transcriptase ribozyme - PubMed (nih.gov)
2019: polymerase η (eta): Human DNA polymerase η has reverse transcriptase activity in cellular environments - PubMed (nih.gov)
2021: Polymerase θ (theta): Polθ reverse transcribes RNA and promotes RNA-templated DNA repair - PubMed (nih.gov)
Erinnern wir uns an den historischen Exkurs über Insertionsmutagenese. Da weiß man welche DNA-Sequenzen man verwendet hat, um seine Experimente durchzuführen. Man weiß, wonach man zu suchen hat.
Bei der Plörre ist die Situation ein wenig komplexer. Es kann sowohl das komplette Plasmid integriert haben[18], als auch linearisierte DNA-Sequenzen[19] insertiert haben
„Bei der Gentherapie stellt die potenzielle Integration des therapeutischen Transgens in das Genom der Wirtszelle ein ernsthaftes Risiko dar, das zu Insertionsmutagenese und Tumorentstehung führen kann. Virale Vektoren werden häufig als Vehikel für die Genübertragung verwendet, sind aber anfällig für Integrationsereignisse.“ (In gene therapy, potential integration of therapeutic transgene into host cell genomes is a serious risk that can lead to insertional mutagenesis and tumorigenesis. Viral vectors are often used as the gene delivery vehicle, but they are prone to undergoing integration events.) [20]
Und das ist nicht neu, das steht sogar im Thermofischer Katalog:
„Obwohl zirkuläre DNA für die transiente Transfektion bevorzugt wird, sind lineare DNA-Vektoren für die Durchführung stabiler Transfektionen effizienter. Dies liegt an der besseren Fähigkeit der linearen DNA, sich in das Wirtsgenom zu integrieren. Obwohl lineare DNA von den Zellen weniger effizient aufgenommen wird, ist lineare DNA mit freien Enden rekombinanter und wird mit größerer Wahrscheinlichkeit in das Wirtschromosom integriert, um stabile Transformanten zu erzeugen.“ (Although circular DNA is preferred for transient transfection, linear DNA vectors are more efficient for performing stable transfections. This is due to the enhanced ability of linear DNA to integrate into the host genome. Specifically, even though linear DNA is taken up by cells less efficiently, linear DNA with free ends is more recombinogenic and more likely to be integrated into the host chromosome to yield stable transformants.) [21]
ThermoFischer ist einer der Lieferanten von BioNTech[22]. Vielleicht hätten die Ugur ja mal einen Katalog schicken können und an der entsprechenden Stelle einen Post-it setzen können?
Und hier schließt sich der Kreis mit einem Zitat aus dem gerade erwähnten Nature portfolio Paper zur Insertionsmutagenese. Also das, was ich schon vor Monaten in meinem Substack geschrieben habe[23], aber mir hört keiner zu. Nein, man muss es erst in Nature lesen, damit man es glaubt. Das ist in der Wissenschaft genauso wie im normalen Leben: Es muss im Mainstream stehen, sonst glaubt man es nicht.
Komplex wird die Angelegenheit nun dadurch, dass wir es nicht nur mit linearisiertem, komplettem Plasmid zu tun haben, das einfach zu finden wäre. Nein, wir haben es leider auch mit diversen Bruchstücken dieses Plasmids zu tun, die von unterschiedlichster Länge und unterschiedlichster Sequenz sein können. Das erschwert das Auffinden der Bruchstücke, weil das Fragment, das man als Nachweisziel auserkoren hat, nicht unbedingt in der betroffenen Krebszelle enthalten sein muss. Besonders bei nur kleinen insertierten Fragmenten wird es schwierig. Da bleibt einem eigentlich nur noch eine komplette Genomsequenzierung, um nach Inserts zu suchen. Das ist heutzutage nicht mehr soooooooooooo teuer, aber billig ist das nicht und Routine schon gar nicht. Also noch nicht. Das kann sich aber ändern, wenn man anfängt das als Beweis vor Gericht einzusetzen. Da zahlt dann in Zukunft die Rechtsschutzversicherung vielleicht für die Erzeugung von Forschungsdaten, die man sich sonst nie leisten könnte. Zugleich mappt man damit das Genom und identifiziert Onkogene und Tumorsuppressorgene auf kosten der Versicherungen. Das setzt aber voraus, dass diese Gutachten auch gesammelt und wissenschaftlich ausgewertet werden.
=== Das SV40 Problem ===
Nun haben wir noch das „kleine“ Problem des SV40 Promotors. Ein Promotor ist in er Genetik ein essentieller Bestandteil eines Gens, der das Ablesen dieses Gens ermöglicht. Der Promotor liegt vor dem Gen. Der Promotor sagt der RNA-Polymerase (Enzym, welches DNA in RNA übersetzt), wo sie sich draufsetzen soll, um mit der Arbeit zu beginnen. Man kann noch Zusatzinformationen hinzufügen darüber, dass viel produziert werden soll mit Enhancer Sequenzen, oder selten abgelesen werden soll mit Silencer Sequenzen. Ratet man, was BioNTech/Pfizer wohl gemacht haben?! Enhancer oder Silencer?
Es gibt viele hunderte bis tausende Promotorsequenzen. Ich bezweifle, dass die Biologen diese Sequenzen wirklich im Detail verstanden haben. Im Labor nimmt man meistens jene, die der Doktorvater mitgebracht hat. So habe ich das gemacht. Man arbeitet, zumindest an der Uni, meist mit erprobten, bewährten Erbstücken der Vorgänger, deren Tücken der Doktorvater kennt, weil er sie selbst gebaut hat.
In der Industrie kann das anders sein. Undefinierte Erbstücke können da regulatorisch zu einem Problem werden. Daher kauft man lieber kommerzielle, genormte Plasmide. Das scheinen BioNTech/Pfizer gemacht zu haben. Deren Plasmid scheint wohl von Agilent zu sein laut USC Professor Dr. Phillip Buckhault[24]. Das bedeutet aber auch, dass man dieses Plasmid nicht unbedingt versteht und was es tut. Man schaut nur, ist ein Promotor drinnen, vertraut dem Hersteller und kennt die Tücken des Plasmids aber nicht unbedingt, wenn man aus der Prokaryotengenetik (Genetik der Bakterien) kommt und immer mit Derivaten von pBR322[25] gearbeitet hat. SV40 ist aber ein Promotor aus dem Simian-Virus 40 (SV40) also aus einem Affenvirus. Dieser Promotor wird somit hauptsächlich von Eukaryoten abgelesen, also Lebewesen mit Zellkern. Wenn man, wie die Chefin der RNA-Bibliothek von BioNTech, aus der Prokaryotengenetik kommt, genau gesagt, seine Promotion auf Cyanobakterien gemacht hat, ist ein Promotor wie der andere, Hauptsache er funktioniert. Dass diese Sequenz Wissenschaftlern, die mit humaner Zellkultur gearbeitet haben, die Haare zu Berge stehen lässt, weil er dafür bekannt ist, dass er das Plasmid direkt in den Zellkern katapultiert[26] und Zellen unsterblich[27] macht, was ein euphemistischer Begriff für „zu Krebszellen[28] macht“ ist, ist einem dabei NICHT bewusst. Ich wusste es auch nicht, denn ich komme aus der Prokaryotengenetik mit E.Coli und den üblichen pBR322 Plasmiden. Ich habe auch erst von anderen Wissenschaftlern erklärt bekommen müssen, dass SV40 eine echt schlechte Idee war. Dabei steht das alles sogar in der Wikipedia[29], wo ich die entsprechenden Paper und Belege herauskopiert habe. Schon wo diese Paper erschienen sind, in einem Journal namens „Oncogene“, müsste eigentlich ausreichen, dass man von dieser Promotorsequenz die Finger lässt.
Wenn, ja wenn man den Promotor vorher googelt und nicht einfach nur schaut, wo man sein Konstrukt laut Klonierungsanweisung hineinbaut. Ich vermute, der Vektor war Teil eines kommerziellen Klonierungskits, die man in der Industrie so gerne verwendet. Da weiß man genetisch gar nicht, was man tut. Man folgt nur den Anweisungen auf dem Beipackzettel und schaut sich nicht einmal die genetische Karte des Plasmids an. Man vertraut dem, was auf dem Klonierungskit steht und wozu der Vektor in der industriellen Produktion regulatorisch für klinische Anwendungen freigegeben ist. pBR322 wäre z. Bsp. NICHT für klinische Anwendungen freigegeben (For research use only. Not intended for any clinical use) und hätte somit nicht verwendet werden dürfen, wäre aber womöglich deutlich sicherer gewesen.
Die Uni Rochester hat da ein paar lustige Versuche gemacht[30]. Dabei zeigte sich, dass Plasmide mit SV40-Promotor die Blutgefäße von Ratten transfizierten, ohne diesen Promotor passierte nichts. Das liegt daran, dass Blutgefäßzellen sich nicht mehr teilen. Plasmide integrieren aber normalerweise nur in sich teilende Zellen. Mit SV40 Promotor erwischt man noch zusätzlich die Zellen, die sonst davongekommen wären, weil sie sich eben nicht mehr teilen.
Das ist aber, wie gesagt, relativ egal, weil auch linearisierte DNA-Fragmente in 10-20% der Fälle[31] die Zellen transfizieren und man es nicht nur mit Plasmiden zu tun hat.
Mit dieser Kombination aus linearisierter DNA mit ungeblockten Enden (weil Bruchstücke) + Plasmid mit SV40 Promotor ist praktisch sichergestellt, dass es JEDEN Zelltyp, ob sich teilend oder nicht mehr teilend, transfiziert und maximal möglicher Schaden angerichtet wird.
Moderna hat KEINEN SV40 Promotor. Sich nicht teilende Zellen müssten im Falle von Moderna-Plörre wohl unbeschadet davongekommen sein, da hat es „nur“ sich teilende Zellen erwischt.
=== Warum die Frage nach der Insertion ins Genom letztendlich leider eigentlich müßig ist ===
Die Diskussion, ob die Plörre nun integriert oder nicht ist aber leider eigentlich müßig, denn leider, leider müssen die Fragmente nicht einmal ins Genom integrieren um Chaos zu stiften, es reicht schon die richtige Sequenz und ihre bloße Anwesenheit um großen Schaden OHNE INSERTION anzurichten. DAS hat leider bisher kaum jemand auf dem Schirm, nicht einmal Jikkyleaks (OK, jetzt schon, ich habe ihn darauf hingewiesen[32]).
====dsRNA ====
Hier hätten wir zwei weitere Problemzonen. Zum einen mach die in der Produktion verwendete T7 RNA Polymerase auch gerne mal doppelsträngige RNA (dsRNA). Es gab 2022 ein Nature Paper, dass man da wohl nun eine T7 Polymerase entwickelt hat, die weniger stressig ist[33], die hat man 2020/21 aber höchstwahrscheinlich nicht verwendet, außer man hatte einen Fluxkompensator und die Ambition ein sicheres Produkt auf dem Markt zu bringen, wovon ich nicht ausgehe.
Warum ist dsRNA ein Problem?
Doppelsträngige RNA (dsRNA) wird in einer Technik namens RNA-Interferenz (RNAi) zum Abschalten von Genen verwendet. Das weiß man sogar als Student, weil es dazu Lernvideos gibt[34]. Doppelsträngige RNA wäre also in der Plörre STRIKT zu vermeiden, weil damit zufällig die Produktion irgendwelcher lebenswichtiger Proteine zufällig abgeschaltet werden könnte. Wie hoch der dsRNA-Anteil in der Plörre ist, ist mir aktuell nicht bekannt. Welche Gene/Proteine betroffen sein könnten ist mir auch unbekannt. Da sowohl Moderna als auch BioNTech/Pfizer ihre Konstrukte unterschiedlich „optimiert“ haben, unterscheidet sich das Schadpotential auf Sequenzebene zudem noch zwischen den beiden Herstellern.
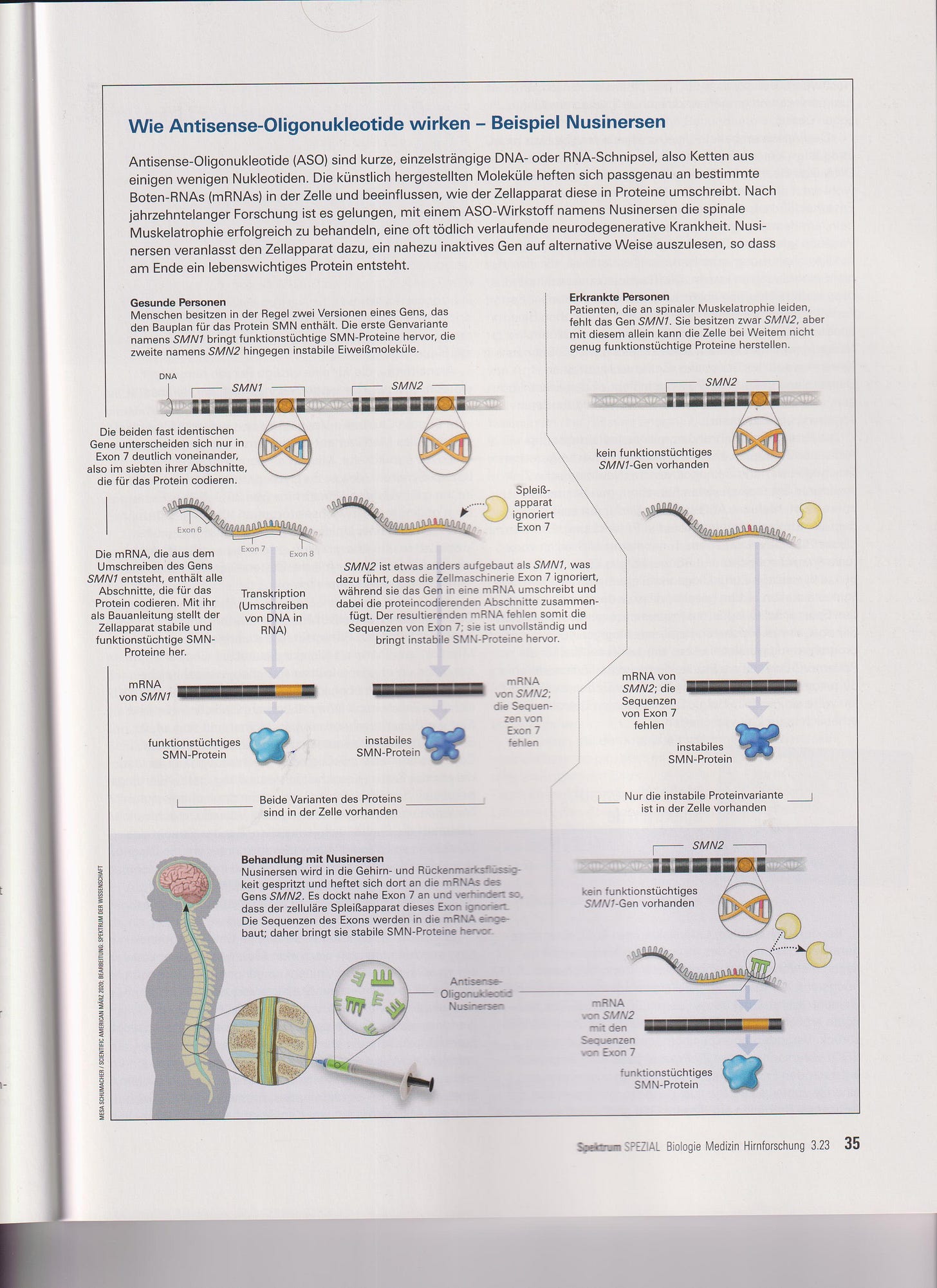
==== Antisense Oligonucleotide ====
Beim Thema Antisense Oligonucleotide mache ich es mir einfach und zitiere aus einem aktuellen Spektrum Heftchen, weil die das schon so schön einfach formuliert haben[35]. Holt euch das Heft am Bahnhofskiosk, wo es auch die andere Spektrum Magazine gibt (und nein, ich bekomme für diesen Werbeblock kein Geld).
„Antisense-Oligonicleotide (ASO) sind kurze, künstlich hergestellte DNA- oder RNA- Stücke. […] ASO werden so konstruiert, dass sie in die Aktivität eines ganz bestimmten Gens eingreifen. Das geschieht, indem sie sich an dessen mRNA heften, indem sie komplementär zu diesem aufgebaut sind. […] Jenachdem, an welchen ihrer Abschnitte da ASO koppelt, produziert die Zelle mehr oder weniger von dem entsprechenden Eiweiß. Damit lassen sich Krankheiten behandeln, die auf einem Mangel oder einem Überschuss bestimmter Proteine beruhen.“
Blöd halt, wenn die Proteinproduktion eines zufälligen anderen Proteins, bei vorher gesunden Menschen hoch- oder runtergefahren würde und damit gegen die „Life on the edge theorie[36] [37]“ verstoßen wird. Dann flockt da unter Umständen eine Menge Protein aus oder es fehlt plötzlich ein lebenswichtiges Eiweiß.
Was bei Krankheiten wie gute Nachrichten klingt, hört sich bei unkontrolliertem Einsatz eher wie eine Drohung an.
„Die Antisense-Oligonukleotide können die Proteinexpression modulieren, indem sie auf hnRNAs oder mRNAs abzielen und eine Interferenz mit dem Spleißen, dem mRNA-Abbau oder einem Translationsstopp bewirken, was schließlich zur Rettung oder Reduzierung der Expression des Zielproteins führt.“ (The antisense oligonucleotides can modulate the protein expression via targeting hnRNAs or mRNAs and inducing interference with splicing, mRNA degradation, or arrest of translation, finally, resulting in rescue or reduction of the target protein expression.)[38]
Muss man nicht im Detail verstehen, will man nicht unkontrolliert ablaufen haben, solange man gesund ist und das nur Schaden anrichten kann.
Das heißt NICHT, dass das bei den Plörrefragmenten passieren muss. Es ist aber eine Möglichkeit, die nicht ausgeschlossen wurde und billigend in Kauf genommen wurde.
Fazit:
Integration von Plörrebestandteilen ist nicht das einzige Problem mit dem wir konfrontiert sind. Es wurde über die diversen Fragmente an DNA und RNA möglicherweise in zelluläre Regulationsmechanismen eingegriffen, die wir weder kennen und erst recht nicht verstehen.
Dieses Problem war bekannt und vorhersehbar und wurde bewusst ignoriert.
Update 21.11.2023 Spermienvermittelter Gentransfer
Ein möglicherweise übersehenes Problem ist spermienvermittelter Gentransfer. Das Produkt ist über mehrere Wochen als modRNA im Blut nachweisbar. Die Plörre geht in die Gonaden, sie könnte also im Ejakulat mit enthalten sein und an die Spermien binden. Es ist auch denkbar, dass Eizellen direkt transfiziert werden, da das modRNA Produkt auch in die Eierstöcke geht.
An dieser Stelle übersetze und sichere ich nur den Wipedia Artikel zu diesem Thema mit google translate ins Deutsche, da es scheints keine deutsche Version gibt. Scheint ein eher seltener Vorgang zu sein, daher keine akute Gefahr. Dennoch eine nicht auszuschließende Möglichkeit, die nicht getestet wurde. Die Entscheidung von Frauen mit Kinderwunsch nach ungeimpften Männern zu suchen, ist daher mehr als verständlich.
Auch bei diesem Mechanismus muss die modRNA zunächst nicht ins Genom integrieren, sie bindet erst einmal nur an den Kopf des Spermiums um wird dann bei der Verschmelzung zur Zygote mit in die Eizelle transportiert.
Dr Arne Burkhardt - Sperm Replaced By Spike Proteins - Irgendwie muss das Produkt dann wohl in die Hoden gekommen sein?
Der spermienvermittelte Gentransfer (SMGT) ist eine transgene Technik, die Gene auf der Grundlage der Fähigkeit von Spermien überträgt, sich spontan an exogene DNA zu binden, diese zu internalisieren und sie während der Befruchtung in eine Eizelle zu transportieren, um genetisch veränderte Tiere zu produzieren . 1 Unter exogener DNA versteht man DNA, die außerhalb des Organismus entsteht. Mit SMGT wurden transgene Tiere gewonnen, die Effizienz dieser Technik ist jedoch gering. Die geringe Effizienz ist hauptsächlich auf die geringe Aufnahme exogener DNA durch die Spermien zurückzuführen, wodurch die Wahrscheinlichkeit einer Befruchtung der Eizellen mit transfizierten Spermien verringert wird. 2 Um durch SMGT erfolgreich transgene Tiere zu produzieren, müssen die Spermatozoen die exogene DNA im Kopf anheften und diese transfizierten Spermatozoen müssen ihre Funktionalität zur Befruchtung der Eizelle beibehalten. 2 Von SMGT produzierte gentechnisch veränderte Tiere sind für die Forschung in biomedizinischen, landwirtschaftlichen und veterinärmedizinischen Bereichen nützlich. SMGT könnte auch bei der Generierung von Tieren als Modelle für menschliche Krankheiten nützlich sein oder zu zukünftigen Entdeckungen im Zusammenhang mit der Gentherapie beim Menschen führen.
Spermienvermittelter Gentransfermechanismus
Die SMGT-Methode nutzt die Samenzelle, einen natürlichen Vektor genetischen Materials, um exogene DNA zu transportieren. Die exogenen DNA-Moleküle binden an die Zellmembran des Samenzellenkopfes. Diese Bindung und Internalisierung der DNA ist kein zufälliges Ereignis. Die exogene DNA interagiert mit den DNA-bindenden Proteinen (DBPs), die auf der Oberfläche der Samenzelle vorhanden sind. 3 Spermatozoen werden auf natürliche Weise durch einen in der Samenflüssigkeit von Säugetieren vorhandenen Hemmfaktor vor dem Eindringen exogener DNA-Moleküle geschützt. Dieser Faktor blockiert die Bindung von Spermien und exogener DNA, da DBPs in Gegenwart des Hemmfaktors ihre Fähigkeit verlieren, an exogene DNA zu binden. Fehlt dieser Hemmfaktor, können DBPs auf Spermien mit der DNA interagieren und diese dann in die Zelle transportieren. Daher muss die Samenflüssigkeit unmittelbar nach der Ejakulation durch ausgiebiges Waschen aus den Spermienproben entfernt werden . 3 Nach der Internalisierung der DNA muss die exogene DNA in das Genom integriert werden. Es werden verschiedene Mechanismen für die DNA-Integration vorgeschlagen, darunter die Integration von DNA bei der Aktivierung der Eizelle, bei der Kerndekondensation oder bei der Bildung der Vorkerne. Alle diese vorgeschlagenen Mechanismen implizieren jedoch, dass die Integration der DNA nach dem Eindringen der Samenzelle in die Eizelle erfolgt. 3
Kontroverse um den durch Spermien vermittelten Gentransfer
Der durch Spermien vermittelte Gentransfer gilt als umstritten, da er sich trotz der Erfolge noch nicht als zuverlässige Form der Genmanipulation etabliert hat. Skepsis entsteht aufgrund der Annahme, dass es zu einem evolutionären Chaos kommen könnte, wenn Spermien als Vektoren für exogene DNA fungieren könnten. 4 Eine vernünftige Annahme besagt, dass Spermien äußerst resistent gegen das Risiko sein sollten, exogene DNA-Moleküle aufzunehmen, da der Fortpflanzungstrakt freie DNA-Moleküle enthält. SMGT wurde experimentell nachgewiesen und folgte der Annahme, dass die Natur Barrieren gegen SMGT aufweist. Diese Barrieren sind nicht immer absolut und könnten die inkonsistenten experimentellen Ergebnisse von SMGT erklären. 4 Wenn es natürliche Barrieren gegen SMGT gibt, dann handelt es sich bei den Erfolgen möglicherweise nur um ungewöhnliche Fälle, in denen die Barrieren versagt haben. Es wurden zwei Hindernisse identifiziert; Der Hemmfaktor in der Samenflüssigkeit, der die Bindung an fremde DNA-Moleküle verhindert, und eine endogene Spermien-Nukleaseaktivität, die bei der Interaktion von Spermien mit fremden DNA-Molekülen ausgelöst wird. 4 Diese Schutzmaßnahmen geben Anlass zu der Annahme, dass unbeabsichtigte Interaktionen zwischen Spermien und exogenen genetischen Sequenzen auf ein Minimum beschränkt werden. Diese Barrieren bieten Schutz vor der Gefahr, dass jede Befruchtung zu einem potenziell mutagenen Ereignis werden könnte. 4
Anwendungen des Spermien-vermittelten Gentransfers
Tiertransgenese
Transgene Tiere wurden erfolgreich mithilfe von Gentransfertechniken wie dem Spermien-vermittelten Gentransfer hergestellt. Obwohl diese Produktion erfolgreich war, ist die Effizienz des Prozesses gering. Die geringe Effizienz von SMGT bei der Produktion transgener Tiere ist hauptsächlich auf die schlechte Aufnahme der exogenen DNA durch die Spermien zurückzuführen, wodurch die Anzahl der befruchteten Eizellen mit transfizierten Spermien verringert wird. 5 Von 1989 bis 2004 gab es über 30 Anträge auf die Produktion lebensfähiger transgener Tiere mittels SMGT, aber nur etwa 25 Prozent davon wiesen eine Übertragung der Transgene über die F 0 -Generation hinaus nach . 4 Diese Übertragung ist erforderlich, um eine nutzbare Tiertransgenese zu beanspruchen. Früheren Studien zufolge erwiesen sich zahlreiche Tierarten, darunter Säugetiere, Vögel, Insekten und Fische, als anfällig für SMGT-Techniken, was darauf hindeutet, dass SMGT eine breite Anwendbarkeit bei einer Vielzahl metazoischer Arten aufweist. 4 Trotz der geringen Häufigkeit der Übertragung von Transgenen lag die Häufigkeit von Phänotypveränderungen und der gesamten Tiertransgenese in einigen Experimenten derzeit bei bis zu 80 Prozent. 4
Gentherapie
Der potenzielle Einsatz des durch Spermien vermittelten Gentransfers für die somatische Gentherapie von Embryonen ist eine Möglichkeit für zukünftige Forschung. Eine embryonale somatische Gentherapie wäre von Vorteil, da offenbar ein umgekehrter Zusammenhang zwischen dem Alter des Patienten und der Wirksamkeit der Gentherapie besteht. Ideal wäre daher die Möglichkeit einer gentherapeutischen Behandlung, bevor irreversible Schäden entstehen. 4 Ein Großteil der Experimente, die über eine erfolgreiche SMGT berichten, liefert Hinweise auf die Übertragung und Aufrechterhaltung von Transgenen nach der Befruchtung. 6 SMGT bietet potenzielle Vorteile als einfache und kostengünstige Methode der Gentherapie, insbesondere im Gegensatz zur pronukleären Mikroinjektion, einer anderen transgenen Technik. Dennoch hat sich SMGT trotz einiger Erfolge und seines potenziellen Nutzens noch nicht als zuverlässige Form der genetischen Veränderung etabliert. 6
Update:
16.09.2024: A reverse transcriptase ribozyme - PubMed (nih.gov), Human DNA polymerase η has reverse transcriptase activity in cellular environments - PubMed (nih.gov), Polθ reverse transcribes RNA and promotes RNA-templated DNA repair - PubMed (nih.gov)
Verweise
1. Lavitrano M, Giovannoni R, Cerrito MG. 2013. Methoden für den Spermien-vermittelten Gentransfer. Methoden Molekularbiologie. 927:519-529.
2. García-Vázquez FA, Ruiz S, Grullón LA, Ondiz AD, Gutiérrez-Adán A, Gadea J. 2011. Faktoren, die den durch Schweinespermien vermittelten Gentransfer beeinflussen. Forschung in der Veterinärwissenschaft. 91(3):446-53.
3. Lavitrano M, Busnelli M, Cerrito MG, Giovannoni R, Manzini S, Vargiolu A. 2006. Spermienvermittelter Gentransfer. Fortpflanzung, Fruchtbarkeit und Entwicklung. 18:19-23.
4. Smith K, Spadafora C. 2005. Spermienvermittelter Gentransfer: Anwendungen und Implikationen. BioEssays. 27(5):551-562.
5. Collares T, Campos VF, de Leon PM, Moura, Cavalcanti PV, Amaral, MG, et al. 2011. Transgenübertragung bei Hühnern durch Spermien-vermittelten Gentransfer nach Entfernung des Samenplasmas und Behandlung exogener DNA mit Dimethylsulfoxid oder N,N-Dimethylacetamid. Zeitschrift für Biowissenschaften. 36(4):613-620.
6. Smith K. 2004. Gentherapie: die potenzielle Anwendbarkeit der Gentransfertechnologie auf die menschliche Keimbahn. Internationale Zeitschrift für medizinische Wissenschaften. 1(2):76-91.
[1] Lindenmaier W, Nguyen-Huu MC, Stratmann LM, Blin N, Wurtz T, Hauser HJ, Giesecke K, Land H, Jeep S, Grez M, Sippel AE, Schütz G. The isolation and characterization of the chicken lysozyme and ovomucoid gene. J Steroid Biochem. 1980 Jan;12:211-8. doi: 10.1016/0022-4731(80)90270-8. PMID: 6252379.
[2] Sippel AE, Land H, Lindenmaier W, Nguyen-Huu MC, Wurtz T, Timmis KN, Giesecke K, Schütz G. Cloning of chicken lysozyme structural gene sequences synthesized in vitro. Nucleic Acids Res. 1978 Sep;5(9):3275-94. doi: 10.1093/nar/5.9.3275. PMID: 568256; PMCID: PMC342248.
[3] Ranzani M, Annunziato S, Adams DJ, Montini E. Cancer gene discovery: exploiting insertional mutagenesis. Mol Cancer Res. 2013 Oct;11(10):1141-58. doi: 10.1158/1541-7786.MCR-13-0244. Epub 2013 Aug 8. PMID: 23928056; PMCID: PMC3836224.
[4] Ärzteblatt DÄGRD. Gentherapie: Nach erstem Todesfall müssen „alle Fakten auf den Tisch“. Deutsches ÄRzteblatt. https://www.aerzteblatt.de/archiv/19705/Gentherapie-Nach-erstem-Todesfall-muessen-alle-Fakten-auf-den-Tisch. Published November 5, 1999.
[5] Wold WS, Toth K. Adenovirus vectors for gene therapy, vaccination and cancer gene therapy. Curr Gene Ther. 2013 Dec;13(6):421-33. doi: 10.2174/1566523213666131125095046. PMID: 24279313; PMCID: PMC4507798.
[6] Lee CS, Bishop ES, Zhang R, Yu X, Farina EM, Yan S, Zhao C, Zheng Z, Shu Y, Wu X, Lei J, Li Y, Zhang W, Yang C, Wu K, Wu Y, Ho S, Athiviraham A, Lee MJ, Wolf JM, Reid RR, He TC. Adenovirus-Mediated Gene Delivery: Potential Applications for Gene and Cell-Based Therapies in the New Era of Personalized Medicine. Genes Dis. 2017 Jun;4(2):43-63. doi: 10.1016/j.gendis.2017.04.001. Epub 2017 Apr 27. PMID: 28944281; PMCID: PMC5609467.
[7] Wang Z, Troilo PJ, Griffiths TG 2nd, Harper LB, Barnum AB, Pacchione SJ, Pauley CJ, Lebron JA, Wolf J, Ledwith BJ. Characterization of integration frequency and insertion sites of adenovirus DNA into mouse liver genomic DNA following intravenous injection. Gene Ther. 2022 Jun;29(6):322-332. doi: 10.1038/s41434-021-00278-2. Epub 2021 Aug 18. PMID: 34404916.
[8] Lee CS, Bishop ES, Zhang R, Yu X, Farina EM, Yan S, Zhao C, Zheng Z, Shu Y, Wu X, Lei J, Li Y, Zhang W, Yang C, Wu K, Wu Y, Ho S, Athiviraham A, Lee MJ, Wolf JM, Reid RR, He TC. Adenovirus-Mediated Gene Delivery: Potential Applications for Gene and Cell-Based Therapies in the New Era of Personalized Medicine. Genes Dis. 2017 Jun;4(2):43-63. doi: 10.1016/j.gendis.2017.04.001. Epub 2017 Apr 27. PMID: 28944281; PMCID: PMC5609467.
[9] Zhang L, Richards A, Khalil A, Wogram E, Ma H, Young RA, Jaenisch R. SARS-CoV-2 RNA reverse-transcribed and integrated into the human genome. bioRxiv [Preprint]. 2020 Dec 13:2020.12.12.422516. doi: 10.1101/2020.12.12.422516. PMID: 33330870; PMCID: PMC7743078.
[10] Anandamide. Pfizer and Moderna bivalent vaccines contain 20-35% expression vector and are transformation competent in E.coli. Substack. https://anandamide.substack.com/p/pfizer-and-moderna-bivalent-vaccines. Published March 8, 2023.
[11] Anandamide. Failure of the linearization reaction in the Pfizer bivalent vaccine manufacturing process. Substack. https://anandamide.substack.com/p/failure-of-the-linearization-reaction. Published March 14, 2023.
[12] Anandamide. DNA contamination in Pfizer monovalent vaccines. Substack. https://anandamide.substack.com/p/dna-contamination-in-pfizer-monovalent. Published March 25, 2023.
[13] Anandamide. DNA contamination in 8 vials of Pfizer monovalent mRNA vaccines. Substack. https://anandamide.substack.com/p/dna-contamination-in-8-vials-of-pfizer. Published March 29, 2023.
[14] Anandamide. Independent Sanger Sequencing verification of plasmid amplicons in BNT162b2. Substack. https://anandamide.substack.com/p/independent-sanger-sequencing-verification. Published June 23, 2023.
[15] Anandamide. BNT162b2 vials tested in South Carolina deliver qPCR CTs in the 18-19 range. Substack. https://anandamide.substack.com/p/bnt162b2-vials-tested-in-south-carolina. Published July 12, 2023.
[16] Anandamide. South Carolina Senate hearing. By Anandamide. https://anandamide.substack.com/p/south-carolina-senate-hearing. Published September 18, 2023.
[17] https://t.me/DrBines_verbales_Vitriol/1430
[18] Haraguchi T, Koujin T, Shindo T, Bilir Ş, Osakada H, Nishimura K, Hirano Y, Asakawa H, Mori C, Kobayashi S, Okada Y, Chikashige Y, Fukagawa T, Shibata S, Hiraoka Y. Transfected plasmid DNA is incorporated into the nucleus via nuclear envelope reformation at telophase. Commun Biol. 2022 Jan 20;5(1):78. doi: 10.1038/s42003-022-03021-8. PMID: 35058555; PMCID: PMC8776997.
[19] Lim S, Yocum RR, Silver PA, Way JC. High spontaneous integration rates of end-modified linear DNAs upon mammalian cell transfection. Sci Rep. 2023 Apr 26;13(1):6835. doi: 10.1038/s41598-023-33862-0. PMID: 37100816; PMCID: PMC10133325.
[20] Lim S, Yocum RR, Silver PA, Way JC. High spontaneous integration rates of end-modified linear DNAs upon mammalian cell transfection. Sci Rep. 2023 Apr 26;13(1):6835. doi: 10.1038/s41598-023-33862-0. PMID: 37100816; PMCID: PMC10133325.
[21] Plasmid transfection | Thermo Fisher Scientific - IE. https://www.thermofisher.com/de/de/home/references/gibco-cell-culture-basics/transfection-basics/applications/plasmid-transfection.html.
[22] Vitriol DV. Liste der BioNTech Zulieferer (Work in Progress). Substack. https://drbine.substack.com/p/liste-der-biontech-zulieferer-work. Published May 9, 2023.
[23] Vitriol DV. Ugurs grenzdebile Schwachsinnsideen im Protein Design. Substack. https://drbine.substack.com/p/ugurs-grenzdebile-schwachsinnsideen-f85. Published July 20, 2023.
[24] SC 4 FREEDOM. SC Senate Hearing - USC Professor Dr. Phillip Buckhaults. YouTube. September 2023.
[25] pBR322 vector (VCT1117) - Creative Biogene. https://www.creative-biogene.com/pBR322-vector-VCT1117-392-17.html?msclkid=59a8421c8acf1685c03f2705975e93b1.
[26] Ahuja D, Sáenz-Robles MT, Pipas JM. SV40 large T antigen targets multiple cellular pathways to elicit cellular transformation. Oncogene. 2005 Nov 21;24(52):7729-45. doi: 10.1038/sj.onc.1209046. PMID: 16299533
[27] Toouli CD, Huschtscha LI, Neumann AA, Noble JR, Colgin LM, Hukku B, Reddel RR. Comparison of human mammary epithelial cells immortalized by simian virus 40 T-Antigen or by the telomerase catalytic subunit. Oncogene. 2002 Jan 3;21(1):128-39. doi: 10.1038/sj.onc.1205014. PMID: 11791183.
[28] Lilyestrom W, Klein MG, Zhang R, Joachimiak A, Chen XS. Crystal structure of SV40 large T-antigen bound to p53: interplay between a viral oncoprotein and a cellular tumor suppressor. Genes Dev. 2006 Sep 1;20(17):2373-82. doi: 10.1101/gad.1456306. PMID: 16951253; PMCID: PMC1560412.
[29] Wikipedia-Autoren. Großes T-Antigen. de.wikipedia.org. May 2018. https://de.wikipedia.org/wiki/Gro%C3%9Fes_T-Antigen.
[30] Nuclear targeting of Plasmids and Protein-DNA complexes - Research projects - Dean Lab - University of Rochester Medical Center. https://www.urmc.rochester.edu/labs/dean/projects/nuclear-targeting-of-plasmids-and-protein-dna-comp.aspx.
[31] Lim S, Yocum RR, Silver PA, Way JC. High spontaneous integration rates of end-modified linear DNAs upon mammalian cell transfection. Sci Rep. 2023 Apr 26;13(1):6835. doi: 10.1038/s41598-023-33862-0. PMID: 37100816; PMCID: PMC10133325.
[32] https://twitter.com/Jikkyleaks/status/1708230178151997599
[33] Dousis A, Ravichandran K, Hobert EM, Moore MJ, Rabideau AE. An engineered T7 RNA polymerase that produces mRNA free of immunostimulatory byproducts. Nat Biotechnol. 2023 Apr;41(4):560-568. doi: 10.1038/s41587-022-01525-6. Epub 2022 Nov 10. PMID: 36357718; PMCID: PMC10110463.
[34] RNA Interferenz • Ablauf, Funktion und Anwendung. Studyflix. https://studyflix.de/biologie/rna-interferenz-2557.
[35] Spektrum der Wissenschaft | Spektrum Spezial BMH - Gentherapie. 1. Auflage | 2023 | beck-shop.de. https://www.beck-shop.de/spektrum-der-wissenschaft-spektrum-spezial-bmh-gentherapie/product/36096561.
[36] Vitriol DV. Ugurs grenzdebile Schwachsinnsideen im Protein Design. Susbtack. https://drbine.substack.com/p/ugurs-grenzdebile-schwachsinnsideen-9d9. Published July 18, 2023.
[37] Vendruscolo M, Knowles TP, Dobson CM. Protein solubility and protein homeostasis: a generic view of protein misfolding disorders. Cold Spring Harb Perspect Biol. 2011 Dec 1;3(12):a010454. doi: 10.1101/cshperspect.a010454. PMID: 21825020; PMCID: PMC3225949. https://pubmed.ncbi.nlm.nih.gov/21825020/
[38] Sardone V, Zhou H, Muntoni F, Ferlini A, Falzarano MS. Antisense Oligonucleotide-Based Therapy for Neuromuscular Disease. Molecules. 2017 Apr 5;22(4):563. doi: 10.3390/molecules22040563. PMID: 28379182; PMCID: PMC6154734.



Vielen Dank für hervorragende verständliche Darlegung!
Ich habe mittlerweile Buckhaults gesehen und McKernan. Da frage ich mich, warum keiner von den beiden mal auf den Gedanken gekommen ist ein Taschentuch beiseite zu nehmen und dies intim zu liebkosen. Wir wissen doch, das die Plörre es bis in den Hoden schafft. Falls dort also sich die DNA einbetten sollte, dürfte ein Samenerguß doch ebenfalls ein MIttel zum Nachweis darstellen? Und zumindestens Buckhaults hat zugegeben dreifach gesumpft zu sein.